线粒体DNA作为动物遗传学研究的理想分子标记,被广泛应用于水生生物的遗传多样性、物种分析和分子系统进化等方面。线粒体细胞色素b基因(Cyt b)被广泛应用于鱼类群体遗传多样性研究[3⇓-5]、种群遗传结构分析[6⇓-8]和系统发育研究[9]等,也被用于洪泽湖和洞庭湖的河蚬种群遗传多样性研究[10-11]。另外线粒体细胞色素氧化酶Ⅰ(COⅠ)基因和线粒体16S核糖体RNA(16S rRNA)基因也常被用于鱼类的仔稚鱼鉴定[12]、分子系统进化分析[13-14]、物种鉴定[15-16]、遗传多样性分析[17-18]和DNA条形码研究[19⇓-21],此外被广泛应用于贝类遗传多样性分析[22]、系统学研究[23]和种间鉴定[24]等,但在河蚬遗传多样性分析中尚未见报道。
1 材料与方法
1.1 试验材料
分别从闽江的延平河段(Yanping,YP)、闽侯河段(Minhou,MH)、龙祥岛河段(Longxiangdao,LXD)、马尾河段(Mawei,MW)、长乐河段(Changle,CL)和连江河段(Lianjiang,LJ)随机采集30个试验用蚬,共180个个体,采集地点如图1所示。采集到的河蚬样本保存于-20℃冰箱中备用。
图1
图1
闽江6个不同地理种群河取样点
Fig.1
The sampling sites of 6 populations of C. fluminea in Minjiang River
1.2 试验方法
1.2.1 形态学性状测量
随机选取闽江6个河段河蚬群体样品共180个,每个河段30个,用纸巾将河蚬贝壳表面的水分吸干后,使用游标卡尺测量壳长(Shell length,SL)、壳高(Shell height,SH)和壳宽(Shell width,SW),精确到0.01 mm。使用电子天平测量总重(Toll weight,TW)、壳重(Shell weight,SW)和软体重(Soft body weight,SBW),精确到0.01 g。河蚬的外形见图2。
图2
1.2.2 基因组DNA提取
参照TIANamp Marine Animals DNA Kit试剂盒说明书(TIANGEN)的提取方法,每个河段随机选取12个河蚬斧足组织进行DNA提取。将提取到的72个DNA样品进行1%琼脂糖凝胶电泳和纯度检测。
1.2.3 引物合成
从NCBI网站(https://www.ncbi.nlm.nih.gov/)查找河蚬COⅠ、Cyt b和16S rRNA序列,使用Primer Premier 6分别设计COⅠ、Cyt b和16S rRNA序列引物(表1),送至福州擎科生物技术有限公司进行合成。
表1 引物信息
Tab.1
| 基因Gene | 引物序列(5’-3’) Primer sequence (5’-3’) | 序列登记号GenBank |
|---|---|---|
| COⅠ | F:GCTATTCCAGGGACTTTACTA R:CCAGCTAACACAGGCATT | AY097303.1 |
| Cyt b | F:TTATAGGGTCGGCAGGAA R:GCATATTGGTCGTGGTATT | MG546300.1 |
| 16S rRNA | F:TTAACGGCTGCGATTGAA R:CGAACAGTCCTACTATTATACC | NC_046410.1:4439~5453 |
1.2.4 COⅠ、Cyt b和16S rRNA基因片段的PCR扩增和测序
COⅠ、Cyt b和16S rRNA基因片段的PCR扩增反应体系为 40 μL:Premix TaqTM (TaKaRa) 20 μL、上下游引物各1 μL、DNA模板2 μL、去离子水16 μL,扩增程序如表2所示。将PCR产物进行1%凝胶电泳,选取条带单一且符合预期大小片段的PCR产物,送至福州擎科生物技术有限公司进行双末端测序。
表2 PCR扩增程序
Tab.2
| 基因Gene | 预变性 Predenaturation | 单循环步骤Single cycle step | 循环次数 Cycles | 后延伸 Posterior extension | ||
|---|---|---|---|---|---|---|
| 变性Denaturation | 退火Annealing | 延伸Extension | ||||
| COⅠ | 94℃,5 min | 94℃,30 s | 55℃,45 s | 72℃,45 s | 35 | 72℃,10 min |
| Cyt b | 53℃,45 s | |||||
| 16S rRNA | 52℃,30 s | |||||
1.3 数据分析
使用Excel 2007对形态学数据进行初步计算变异系数,再采用SPSS 20.0 软件进行单因素方差分析和显著性分析,P<0.05表示组间差异显著。使用Mega 7软件Clustal W进行多序列比对,人工校对和计算闽江6个不同河段河蚬群体的COⅠ、Cyt b和16S rRNA基因片段序列长度和碱基组成。使用Dnasp 5软件计算闽江6个河段河蚬群体COⅠ、Cyt b和16S rRNA基因的遗传多样性。使用Mega 7软件的最大似然法(Maximum likelihood,ML)和邻接法(Neighbor-joining,N-J)构建闽江6个河段河蚬群体COⅠ、Cyt b和16S rRNA基因片段序列分子系统进化树。通过中性检验Tajima’s D、Fu’s Fs和核苷酸不配对分布图来检测闽江河蚬的群体历史动态。
2 结果与分析
2.1 闽江6个河段河蚬形态学性状分析
闽江6个河段河蚬的形态学性状统计量见表3。不同河段河蚬壳长、壳高和壳宽的变异系数较小,而软体重、壳重和总重的变异系数较大,其中软体重、壳重和总重变异系数最大的群体分别为LJ(53.30%)、MW(48.82%)和MW(49.82%)。LXD群体的壳高/壳长显著高于YP、MW和CL(P<0.05),与MH和LJ的差异不显著(P>0.05)。LJ群体的壳宽/壳长极显著高于YP、MH、LXD、MW(P<0.01),与CL的差异不显著(P>0.05)。MW和LJ群体的软体重/总重均极显著高于YP、MH、LXD、CL(P<0.01),MW和LJ之间的差异不显著(P>0.05)。LJ群体的壳重/总重极显著高于YP、MH和MW(P<0.01),显著高于CL(P<0.05),与LXD的差异不显著(P>0.05)。
表3 闽江6个河段河蚬群体的形态学性状统计量(n=180)
Tab.3
| 性状 Traits | 延平YP | 闽侯MH | 龙祥岛LXD | 马尾MW | 长乐CL | 连江LJ | ||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 平均值± 标准差 Mean±SD | 变异 系数/% CV | 平均值± 标准差 Mean±SD | 变异 系数/% CV | 平均值± 标准差 Mean±SD | 变异 系数/% CV | 平均值± 标准差 Mean±SD | 变异 系数/% CV | 平均值± 标准差 Mean±SD | 变异 系数/% CV | 平均值± 标准差 Mean±SD | 变异 系数/% CV | |
| 壳长/mm SL | 19.32±2.85 | 14.76 | 19.68±1.48 | 7.51 | 21.27±2.13 | 10.018 | 21.41±3.62 | 16.92 | 19.98±3.38 | 16.90 | 18.02±2.66 | 14.78 |
| 壳高/mm SH | 17.46±2.16 | 12.38 | 18.23±1.50 | 8.24 | 19.90±2.13 | 10.679 | 19.40±3.23 | 16.64 | 18.04±2.54 | 14.06 | 16.67±2.66 | 15.95 |
| 壳宽/mm SW | 11.46±1.28 | 11.150 | 12.29±1.01 | 8.25 | 13.09±1.15 | 8.804 | 12.70±1.83 | 14.37 | 12.58±1.37 | 10.86 | 11.80±1.72 | 14.56 |
| 软体重/g SBW | 0.43±0.18 | 42.66 | 0.43±0.11 | 25.18 | 0.60±0.16 | 27.215 | 0.79±0.41 | 52.59 | 0.55±0.29 | 52.78 | 0.56±0.30 | 53.30 |
| 壳重/g SW | 1.50±0.48 | 32.16 | 1.76±0.45 | 25.77 | 2.29±0.58 | 25.335 | 2.09±1.02 | 48.82 | 1.88±0.61 | 32.28 | 1.64±0.71 | 43.03 |
| 总重/g TW | 2.47±0.80 | 32.29 | 2.83±0.67 | 23.76 | 3.62±0.93 | 25.582 | 3.42±1.70 | 49.82 | 3.04±1.13 | 37.04 | 2.51±1.11 | 44.13 |
| 壳高/壳长 SH/SL | 0.91±0.06a | 6.60 | 0.93±0.04ab | 3.99 | 0.94±0.03b | 2.834 | 0.91±0.06a | 6.17 | 0.91±0.04a | 4.71 | 0.92±0.03ab | 2.97 |
| 壳宽/壳长 SW/SL | 0.60±0.07a | 12.24 | 0.63±0.03b | 4.99 | 0.62±0.03ab | 5.043 | 0.60±0.04a | 6.49 | 0.64±0.05bc | 8.33 | 0.66±0.03c | 4.28 |
| 软体重/总重 SBW/TW | 0.17±0.03b | 19.03 | 0.15±0.02a | 9.86 | 0.17±0.02ab | 13.102 | 0.23±0.04c | 17.93 | 0.17±0.04b | 21.64 | 0.22±0.04c | 16.28 |
| 壳重/总重 SW/TW | 0.61±0.07a | 11.82 | 0.62±0.042a | 6.760 | 0.63±0.03ab | 3.897 | 0.61±0.07a | 10.95 | 0.63±0.05a | 8.07 | 0.66±0.03b | 5.09 |
注:同行数据肩标不同字母表示差异显著(P<0.05),相同字母表示差异不显著(P>0.05)。
Notes:In the same row,values with different small letter superscripts meant significant difference (P <0.05),while with the same letter superscripts meant no significant difference (P>0.05).
2.2 闽江6个河段河蚬群体COⅠ、Cyt b和16S rRNA基因的碱基组成
采集闽江6个河段河蚬群体共72个样品进行线粒体CO Ⅰ、Cyt b和16S rRNA基因PCR扩增、测序和分析。使用MEGA7.0对COⅠ、Cyt b和16S rRNA基因序列进行比对、人工校对和剪切后,分别获得444、466、364 bp的序列片段。COⅠ基因中A、C、T和G四种碱基的平均含量分别为32.3%、16.9%、33.5%和17.3%,A+T和C+G的含量分别为65.8%和34.2%;Cyt b基因中A、C、T和G四种碱基的平均含量分别为39.3%、16.0%、32.1%和12.6%,A+T和C+G含量分别为71.4%和28.6%;16S rRNA基因A、C、T和G 4种碱基的平均含量分别为34.6%、15.2%、34.4%和15.7%,A+T和C+G的含量分别为69.1%和30.9%(表4)。闽江6个河段河蚬种群的COⅠ、Cyt b和16S rRNA基因碱基组成基本一致。
表4 闽江6个河段河蚬群体COⅠ、Cyt b和16S rRNA基因碱基组成
Tab.4
| 基因 Gene | 群体 Population | 碱基组成/% Base composition | ||||||
|---|---|---|---|---|---|---|---|---|
| A | C | T | G | A+T | C+G | |||
| COⅠ | 延平 | 36.1 | 18.0 | 29.6 | 16.2 | 65.7 | 34.2 | |
| 闽侯 | 31.2 | 16.5 | 34.6 | 17.6 | 65.8 | 34.1 | ||
| 龙祥岛 | 31.2 | 16.5 | 34.6 | 17.6 | 65.8 | 34.1 | ||
| 马尾 | 32.6 | 17.3 | 32.7 | 17.4 | 65.3 | 34.7 | ||
| 长乐 | 31.2 | 16.6 | 34.6 | 17.6 | 65.8 | 34.2 | ||
| 连江 | 31.2 | 16.5 | 34.6 | 17.6 | 65.8 | 34.1 | ||
| 平均 | 32.3 | 16.9 | 33.5 | 17.3 | 65.8 | 34.2 | ||
| Cyt b | 延平 | 29.0 | 11.4 | 42.1 | 17.5 | 71.1 | 28.9 | |
| 闽侯 | 39.1 | 15.7 | 32.6 | 12.6 | 71.7 | 28.3 | ||
| 龙祥岛 | 40.7 | 16.5 | 30.9 | 11.9 | 71.6 | 28.4 | ||
| 马尾 | 45.4 | 19.3 | 25.5 | 9.8 | 70.9 | 29.1 | ||
| 长乐 | 42.2 | 17.6 | 29.0 | 11.3 | 71.2 | 28.9 | ||
| 连江 | 39.1 | 15.7 | 32.5 | 12.6 | 71.6 | 28.3 | ||
| 平均 | 39.3 | 16.0 | 32.1 | 12.6 | 71.4 | 28.6 | ||
| 16S rRNA | 延平 | 35.3 | 13.6 | 33.9 | 17.1 | 69.2 | 30.7 | |
| 闽侯 | 34.1 | 16.3 | 34.8 | 14.8 | 68.9 | 31.1 | ||
| 龙祥岛 | 34.3 | 16.0 | 34.6 | 15.1 | 68.9 | 31.1 | ||
| 马尾 | 35.0 | 14.5 | 34.2 | 16.3 | 69.2 | 30.8 | ||
| 长乐 | 34.6 | 15.3 | 34.6 | 15.5 | 69.2 | 30.8 | ||
| 连江 | 34.5 | 15.4 | 34.5 | 15.6 | 69.0 | 31.0 | ||
| 平均 | 34.6 | 15.2 | 34.4 | 15.7 | 69.1 | 30.9 | ||
2.3 闽江6个河段河蚬群体COⅠ、Cyt b和16S rRNA基因的的遗传多样性
在COⅠ基因的72条序列中检出20种单倍型即Hap1~Hap20,其中Hap5为YP独享单倍型,Hap6~Hap9为LXD独享单倍型,Hap10~Hap14和Hap16均为MW独享单倍型,Hap17~Hap18为CL独享单倍型,Hap19~Hap20为LJ独享单倍型,其余均为共享型单倍型,如表5所示。6个河段群体的单倍型数量:MW>CL>LXD>YP >LJ >MH,其中Hap3在6个河段群体中分布最多,占比为31.9%(23/72)。在Cyt b基因的71条序列中检出20种单倍型即Ha1~Ha20,其中Ha1、Ha3为YP独享单倍型,Ha8~Ha10为LXD独享单倍型,Ha11~Ha15为MW独享单倍型,Ha16~Ha20为LJ独享单倍型,其余均为共享型单倍型,如表5所示。6个河段群体的单倍型数量:LJ>MW>YP/LXD>CL>MH,其中Ha6在6个河段群体中分布最多,占比为25.4%(18/71)。在16S rRNA基因71条序列中检出9种单倍型即H1~H9,其中H5~H7为LXD独享单倍型,H8~H9为MW独享单倍型,其余均为共享型单倍型,如表5所示。6个河段群体的单倍型数量:LXD>MW/LJ>YP /MH/CL,其中H2在6个河段群体中分布最多,占比为28.2%(20/71)。
表5 闽江6个河段河蚬群体COⅠ、Cyt b和16S rRNA基因单倍型分布
Tab.5
| 基因 Gene | 单倍型 Haplotypes | 延平 YP | 闽侯 MH | 龙祥岛 LXD | 马尾 MW | 长乐 CL | 连江 LJ | 总计 Total | 百分比/% Percentage | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| COⅠ | Hap1 | 4 | 2 | 1 | 7 | 9.7 | ||||||||||||
| Hap2 | 3 | 5 | 3 | 1 | 4 | 16 | 22.2 | |||||||||||
| Hap3 | 3 | 7 | 5 | 2 | 6 | 23 | 31.9 | |||||||||||
| Hap4 | 1 | 2 | 3 | 4.2 | ||||||||||||||
| Hap5 | 1 | 1 | 1.4 | |||||||||||||||
| Hap6 | 1 | 1 | 1.4 | |||||||||||||||
| Hap7 | 1 | 1 | 1.4 | |||||||||||||||
| Hap8 | 1 | 1 | 1.4 | |||||||||||||||
| Hap9 | 1 | 1 | 1.4 | |||||||||||||||
| Hap10 | 2 | 2 | 2.8 | |||||||||||||||
| Hap11 | 1 | 1 | 1.4 | |||||||||||||||
| Hap12 | 1 | 1 | 1.4 | |||||||||||||||
| Hap13 | 3 | 3 | 4.2 | |||||||||||||||
| Hap14 | 1 | 1 | 1.4 | |||||||||||||||
| Hap15 | 1 | 1 | 2 | 2.8 | ||||||||||||||
| Hap16 | 1 | 1 | 1.4 | |||||||||||||||
| Hap17 | 4 | 4 | 5.6 | |||||||||||||||
| Hap18 | 1 | 1 | 1.4 | |||||||||||||||
| Hap19 | 1 | 1 | 1.4 | |||||||||||||||
| Hap20 | 1 | 1 | 1.4 | |||||||||||||||
| 主要单倍型 和占比/% | Hap1 (33.3) | Hap3 (58.3) | Hap3 (41.7) | Hap13 (25.0) | Hap17 (33.3) | Hap3 (50.0) | — | — | ||||||||||
| Cyt b | Ha1 | 4 | 4 | 5.6 | ||||||||||||||
| Ha2 | 4 | 2 | 6 | 8.5 | ||||||||||||||
| Ha3 | 1 | 1 | 1.4 | |||||||||||||||
| Ha4 | 1 | 8 | 9 | 12.7 | ||||||||||||||
| Ha5 | 1 | 3 | 2 | 6 | 8.5 | |||||||||||||
| Ha6 | 8 | 7 | 3 | 18 | 25.4 | |||||||||||||
| Ha7 | 4 | 2 | 3 | 9 | 12.7 | |||||||||||||
| Ha8 | 1 | 1 | 1.4 | |||||||||||||||
| Ha9 | 1 | 1 | 1.4 | |||||||||||||||
| Ha10 | 1 | 1 | 1.4 | |||||||||||||||
| Ha11 | 1 | 1 | 1.4 | |||||||||||||||
| Ha12 | 4 | 4 | 5.6 | |||||||||||||||
| Ha13 | 2 | 2 | 2.8 | |||||||||||||||
| Ha14 | 1 | 1 | 1.4 | |||||||||||||||
| Ha15 | 1 | 1 | 1.4 | |||||||||||||||
| Ha16 | 2 | 2 | 2.8 | |||||||||||||||
| Ha17 | 1 | 1 | 1.4 | |||||||||||||||
| Ha18 | 1 | 1 | 1.4 | |||||||||||||||
| Ha19 | 1 | 1 | 1.4 | |||||||||||||||
| Ha20 | 1 | 1 | 1.4 | |||||||||||||||
| 主要单倍型 和占比/% | Ha1、Ha2 (36.4) | Ha6 (66.7) | Ha6 (58.3) | Ha12 (33.3) | Ha4 (66.7) | Ha6、Ha7 (25.0) | — | — | ||||||||||
| 16S rRNA | H1 | 4 | 4 | 6 | 2 | 16 | 22.5 | |||||||||||
| H2 | 8 | 4 | 6 | 2 | 20 | 28.2 | ||||||||||||
| H3 | 7 | 4 | 4 | 15 | 21.1 | |||||||||||||
| H4 | 5 | 4 | 4 | 13 | 18.3 | |||||||||||||
| H5 | 1 | 1 | 1.4 | |||||||||||||||
| H6 | 1 | 1 | 1.4 | |||||||||||||||
| H7 | 1 | 1 | 1.4 | |||||||||||||||
| H8 | 1 | 1 | 1.4 | |||||||||||||||
| H9 | 3 | 3 | 4.2 | |||||||||||||||
| 主要单倍型 和占比/% | H2 (66.7) | H3 (58.3) | H3、H4 (36.4) | H1、H2 (33.3) | H1、H2 (50.0) | H3、H4 (33.3) | — | — | ||||||||||
在COⅠ、Cyt b和16S rRNA基因中河蚬总的单倍型多样性、核苷酸多样性及平均核苷酸差异数分别为(0.840±0.030)、(0.313±0.008)、136.010,(0.892±0.021)、(0.230±0.001)、98.330和(0.800±0.019)、(0.316±0.009)、108.212(表6)。在COⅠ、Cyt b和16S rRNA基因中单倍型多样性(Hd)最高的群体分别为MW群体、LJ群体和LJ群体,最低的群体分别为MH群体、MH群体和YP群体;核苷酸多样性(π)最高的群体分别为MW群体、LJ群体和LJ群体,最低的群体分别是YP群体、MW群体和YP群体。
表6 闽江6个河段河蚬群体COⅠ、Cyt b和16S rRNA基因的遗传多样性
Tab.6
| 基因 Gene | 群体 Population | 样品数 No.of samples | 单倍型数 No.of haplotypes | 单倍型多样性 Haplotype diversity (Hd±SD) | 核苷酸多样性 Nucleotide diversity (π±SD) | 平均核苷酸差异数 Average number of nucleotide differences (k) |
|---|---|---|---|---|---|---|
| COⅠ | 延平 | 12 | 5 | 0.818±0.070 | 0.303±0.013 | 131.712 |
| 闽侯 | 12 | 2 | 0.530±0.076 | 0.325±0.012 | 141.061 | |
| 龙祥岛 | 12 | 6 | 0.803±0.096 | 0.326±0.012 | 141.364 | |
| 马尾 | 12 | 8 | 0.924±0.057 | 0.341±0.013 | 147.864 | |
| 长乐 | 12 | 7 | 0.879±0.075 | 0.329±0.013 | 142.864 | |
| 连江 | 12 | 4 | 0.682±0.102 | 0.325±0.012 | 141.212 | |
| 合计 | 72 | 20 | 0.840±0.030 | 0.313±0.008 | 136.010 | |
| Cyt b | 延平 | 11 | 5 | 0.782±0.093 | 0.179±0.012 | 76.255 |
| 闽侯 | 12 | 2 | 0.485±0.106 | 0.251±0.012 | 107.152 | |
| 龙祥岛 | 12 | 5 | 0.667±0.141 | 0.213±0.012 | 91.091 | |
| 马尾 | 12 | 6 | 0.848±0.074 | 0.008±0.002 | 3.803 | |
| 长乐 | 12 | 3 | 0.545±0.144 | 0.163±0.012 | 69.455 | |
| 连江 | 12 | 7 | 0.894±0.063 | 0.253±0.012 | 107.985 | |
| 合计 | 71 | 20 | 0.892±0.021 | 0.230±0.001 | 98.330 | |
| 16S rRNA | 延平 | 12 | 2 | 0.485±0.106 | 0.301±0.014 | 103.273 |
| 闽侯 | 12 | 2 | 0.530±0.076 | 0.329±0.014 | 112.955 | |
| 龙祥岛 | 11 | 5 | 0.782±0.093 | 0.339±0.015 | 116.327 | |
| 马尾 | 12 | 4 | 0.773±0.069 | 0.332±0.014 | 113.530 | |
| 长乐 | 12 | 2 | 0.545±0.062 | 0.339±0.014 | 116.182 | |
| 连江 | 12 | 4 | 0.788±0.070 | 0.339±0.014 | 116.424 | |
| 合计 | 71 | 9 | 0.800±0.019 | 0.316±0.009 | 108.212 |
2.4 闽江6个不同河段河蚬群体系统进化树
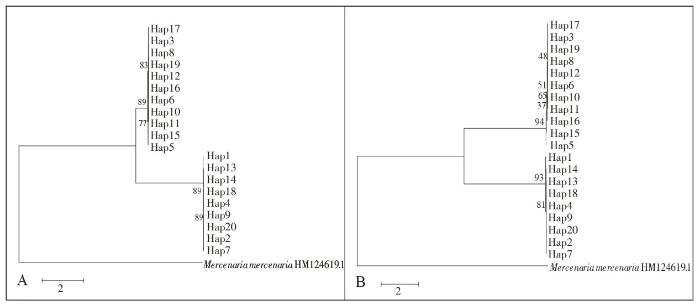
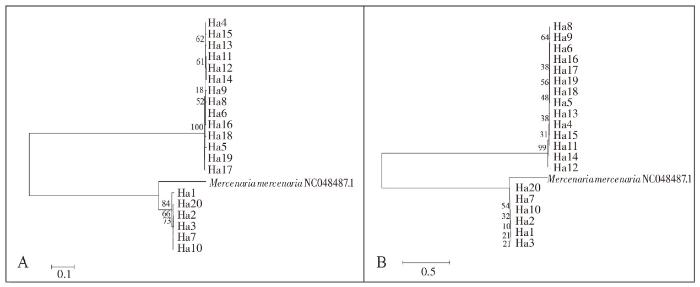
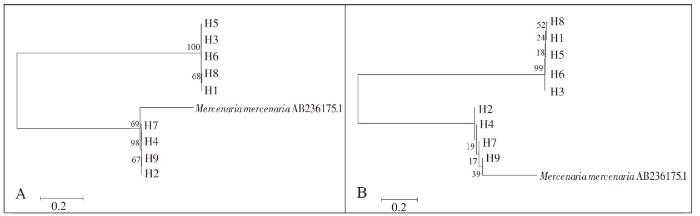
通过ML法和N-J法建立COⅠ、Cyt b和16S rRNA基因系统进化树,以硬壳蛤(Mercenaria mercenaria)作为外类群,bootstrap 1 000。在COⅠ基因ML和N-J系统进化树中均显示,11个单倍型(38个个体)聚为一支,置信度分别为89和94,主要分布在MH、LXD、CL、LJ、MW;9个单倍型(34个个体)聚为另一支,主要分布在YP;20个单倍型聚为一大支后再与硬壳蛤相聚(图3),表明闽江河蚬有2个种且能与硬壳蛤区分开。在Cyt b基因ML和N-J系统进化树中均显示,14个单倍型(49个个体)聚为一支,置信度分别为100和99,主要分布在MW、LJ、CL、LXD、MH;其他6个单倍型(22个个体)与硬壳蛤聚为另一支,主要分布在YP(图4)。在16S rRNA基因ML和N-J系统进化树中均显示,5个单倍体(34个个体)聚为一支,置信度分别为100和99,主要分布在MH、LXD、CL、LJ;另4个单倍体(37个个体)与硬壳蛤聚为另一支,主要分布在YP、MW、CL、LJ(图5)。表明闽江河蚬线粒体基因的进化速度为COⅠ> Cyt b > 16S rRNA。
图3
图3
基于闽江6个河段河洁群体COⅠ基因20个单倍型的最大似然树(ML)(A)和邻接树(N-J)(B)
Fig.3
Maximum likelihood(ML) tree (A) and Neighbor-joining (N-J) tree (B) of 20 haplotypes based on partial col gene sequences of C. fluminea populations in the 6 reaches of Minjiang River
图4
图4
基于闽江6个河段河虮群体Cyt b基因20个单倍型的最大似然树(ML)(A)和邻接树(N-J)(B)
Fig.4
Maximum likelihood (ML) tree(A) and Neighbor-joining (N-J) tree (B) of 20 haplotypes based on partial Cyt b gene sequences of C. fluminea populations in the 6 reaches of Minjiang River
图5
图5
基于闽江6个河段河虮群体16S rRNA基因9个单倍型的最大似然树(ML)(A)和邻接树(N-J)(B)
Fig.5
Maximum likelihood(ML) tree (A) and Neighbor-joining(N-J) tree (B) of 9 haplotypes based on partial 16S rRNA gene sequences of C. fluminea populations in the 6 reaches of Minjiang River
2.5 闽江6个河段河蚬群体历史动态
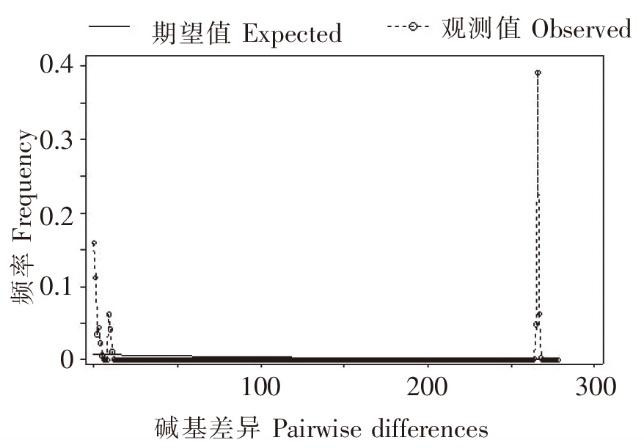
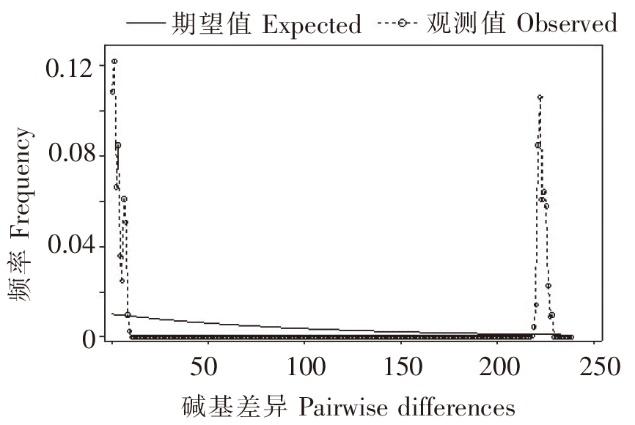
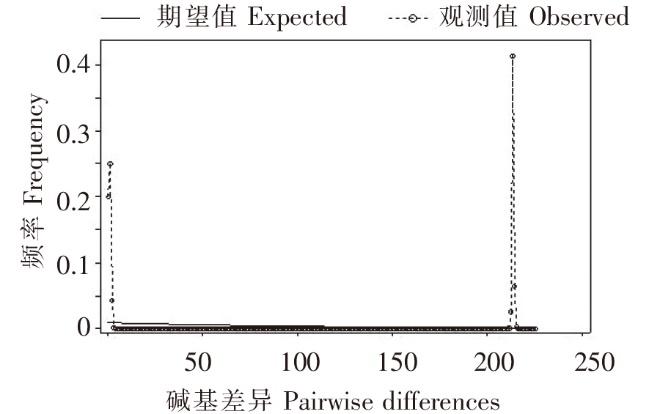
闽江6个河段河蚬群体COⅠ基因的中性检验Tajima’s D值在1.933~2.831之间,其中YP群体差异不显著(P>0.05),其余差异极显著(P<0.01);Fu’s Fs值为10.597~37.394,差异均极显著(P<0.01)。Cyt b基因的中性检验Tajima’s D值在-0.395~2.183之间,其中YP群体和CL群体为负值,其余均为正值;MH群体和LJ群体差异显著(P<0.05),其余差异不显著(P>0.05);Fu’s Fs值在0.303~34.509之间,其中只有MW群体差异不显著(P>0.05),其余均差异极显著(P<0.01)。16S rRNA基因的中性检验Tajima’s D值在2.182~3.042之间,其中YP群体差异显著(P<0.05),其余差异极显著(P<0.01);Fu’s Fs值在17.796~35.355之间,差异均极显著(P<0.01),详见表7。该3个基因的岐点分布图均呈多峰型(图6~图8)。以上均表明闽江河蚬群体很稳定,均未发生过大规模的种群扩张等历史事件。
表7 闽江6个不同河段河蚬COⅠ、Cyt b和16S rRNA基因的中性检验
Tab.7
| 基因 Gene | 群体 Population | Tajima’s D | Fu’s Fs | ||
|---|---|---|---|---|---|
| D | P | Fs | P | ||
| COⅠ | 延平 | 1.933 | >0.10 | 21.049 | 0.000 |
| 闽侯 | 2.831 | <0.001 | 37.394 | 0.000 | |
| 龙祥岛 | 2.791 | <0.001 | 17.454 | 0.000 | |
| 马尾 | 2.593 | <0.01 | 10.597 | 0.000 | |
| 长乐 | 2.470 | <0.01 | 13.791 | 0.000 | |
| 连江 | 2.783 | <0.001 | 26.170 | 0.000 | |
| 合计 | 4.276 | <0.001 | 57.353 | 0.000 | |
| Cyt b | 延平 | -0.240 | >0.10 | 15.064 | 0.000 |
| 闽侯 | 2.183 | <0.05 | 34.509 | 0.000 | |
| 龙祥岛 | 0.996 | >0.10 | 18.303 | 0.000 | |
| 马尾 | 0.183 | >0.10 | 0.303 | 0.249 | |
| 长乐 | -0.395 | >0.10 | 24.696 | 0.000 | |
| 连江 | 2.113 | <0.05 | 12.244 | 0.000 | |
| 合计 | 3.190 | <0.01 | 44.516 | 0.000 | |
| 16S rRNA | 延平 | 2.182 | <0.05 | 34.125 | 0.000 |
| 闽侯 | 2.827 | <0.001 | 35.060 | 0.000 | |
| 龙祥岛 | 2.860 | <0.001 | 17.796 | 0.000 | |
| 马尾 | 2.795 | <0.001 | 24.312 | 0.000 | |
| 长乐 | 3.042 | <0.001 | 35.355 | 0.000 | |
| 连江 | 2.986 | <0.001 | 24.525 | 0.000 | |
| 合计 | 4.861 | <0.001 | — | 0.000 | |
图6
图6
基于CO|基因闽江河芸种群的岐点分布
Fig.6
The mismatch distribution analysis of C. fluminea population based on partialco l gene sequences in Minjiang River
图7
图7
基于Cyt b 基因闽江河觑种群的岐点分布
Fig.7
The mismatch distribution analysis of C. fluminea population based on partial Cyt b gene sequences in Minjiang River
图8
图8
基于16S rRNA基因闽江河种群的岐点分布
Fig.8
The mismatch distribution analysis ofC. fluminea population based on partial 16S rRNA gene sequences in Minjiang River
3 讨论
3.1 形态学差异分析
环境是影响双壳类动物个体差异的主要原因之一,虽然河蚬具有因栖息地环境变化而形成高度形态变异的特性,但是已有的研究表明形态差异很难被用于区分不同河段的河蚬[37⇓-39]。本研究分析了闽江6个河段河蚬群体的形态学数据,结果表明壳长、壳高和壳宽的变异系数较小,而软体重、壳重和总重的变异系数较大,其中软体重的变异系数最大,为53.30%,这与学者对凹线仙女蚬(Cyrenobatissa subsulcata)[37]、波纹巴菲蛤(Paphia undulata)[40]和红树蚬(Polymesoda erosa)[41]的研究结果相一致。综合对比壳高/壳长(LXD>MH>LJ>YP/MW/CL)、壳宽/壳长(LJ>CL>MH>LXD>YP/MW)、软体重/总重(MW>LJ>YP/LXD/CL>MH)和壳重/总重(LJ>LXD/CL>MH>YP/MW)的数据,可以看出YP和MW群体的壳高/壳长、壳宽/壳长和壳重/总重均最小,但MW群体的软体重/总重最大,YP群体的软体重/总重居中;表明闽江6个河段的河蚬在形态上无显著性差异,因此无法通过形态学数据区分闽江不同河段的河蚬。
3.2 遗传多样性与进化速度比较分析
本研究综合分析了闽江6个河段河蚬群体的COⅠ(444 bp)、Cyt b(466 bp)和16S rRNA(364 bp)基因部分序列片段,3个基因中A+T含量(65.8%、71.4%和69.1%)明显高于C+G的含量(34.2%、28.6%和30.9%)。捞刀河和洪泽湖河蚬的线粒体COⅠ基因序列中发现A+T的含量(64.3%和65.0%)高于C+G的含量(35.7%和35.0%)[32,42],且洞庭湖河蚬线粒体Cyt b基因部分序列中也发现A+T的含量(70.4%)明显高于C+G的含量(29.6%)[11],表明了COⅠ和Cyt b基因序列在不同地域的河蚬中具有可比性。16S rRNA基因在其他贝类中也有相似的结果,闫永斌等[22]在日本镜蛤(Dosinia japonica)、四角蛤蜊(Mactra veneriformis)、中国蛤蜊(Mactra chinensis)和西施舌(Coelomactra antiquata)中发现A+T (67.11%、61.35%、59.30%和60.59%)含量高于C+G (32.89%、38.65%、40.70%和39.41%);陈丽梅等[23]在大竹蛏(Solen grandis)、长竹蛏(Solen strictus)和小刀蛏(Cultellus attenuatus)中发现A+T (60.8%、62.3%和62.5%)的含量高于C+G (39.2%、37.7%和37.5%)的含量;孙超等[24]在光滑河蓝蛤(Potamocorbula laevis)、黑龙江河蓝蛤(P.amurensis)、焦河蓝蛤(P.ustulata)河红肉河蓝蛤(P.rubromuscula)中也发现A+T (61.48%、61.48%、61.53%和61.10%)的含量高于C+G (38.52%、38.52%、38.46%和38.90%)的含量。可见(A+T)%含量高于(C+G)%是许多双壳类动物线粒体碱基组成的共性。
单倍型多样性(Hd)和核苷酸多样性(π)是评价生物遗传多样性的两个重要指标[43]。本研究发现,闽江6个河段河蚬群体COⅠ和Cyt b基因的单倍型数量和多样性均大于16S rRNA基因,可见COⅠ和Cyt b基因的单倍型多样性比16S rRNA基因高。COⅠ、Cyt b和16S rRNA基因核苷酸多样性差异不大。以上都表明了COⅠ和Cyt b基因比16S rRNA基因遗传多样性更高。
3.3 群体历史动态分析
中性检验和核苷酸不配对分布图是检验种群历史动态的两种方法。在中性检验中,若Tajima’s D值和Fu’s Fs值均为正值,表明种群趋于稳定,若Tajima’s D值为负,且统计学上达到显著水平,表明该群体偏离中性突变理论模型,有可能受到过选择压力、瓶颈效应的作用或者发生过大规模的群体扩张[44-45]。闽江6个河段河蚬群体COⅠ和16S rRNA基因的中性检验Tajima’s D值和Fu’s Fs值均为正值。Cyt b基因Tajima’s D值中YP群体和CL群体为负值,但统计学上差异不显著(P>0.05);Fu’s Fs检验中6个河蚬群体Fs值均大于零。若核苷酸岐点分布图呈现多峰型,则表明种群呈稳定状态,反之,若呈单峰型,则表明种群历史有扩张现象[31]。闽江6个河段河蚬群体的核苷酸岐点分布图均呈多峰型。以上均表明了闽江河蚬群体很稳定,均未发生过大规模的种群扩张等历史事件。
4 结论
本研究综合分析了闽江6个河段河蚬群体形态学数据和线粒体CO Ⅰ(444 bp)、Cyt b(466 bp)和16S rRNA基因(364 bp)部分序列片段。壳长、壳高和壳宽的变异系数较小,而软体重、壳重和总重的变异系数较大,其中软体重的变异系数最大的群体为LJ(53.30%)。在COⅠ、Cyt b和16S rRNA基因中A+T含量相近且明显高于C+G的含量;COⅠ和Cyt b 基因的单倍型数量明显多于16S rRNA基因;COⅠ和Cyt b基因的遗传多样性比16S rRNA基因高;进化速度是COⅠ>Cyt b>16S rRNA基因。COⅠ、Cyt b和16S rRNA基因的中性检验和岐点分布图均表明了闽江河蚬群体很稳定。综上所述,闽江6个河段河蚬群体的COⅠ、Cyt b基因比16S rRNA基因遗传多样性更高、进化速度更快,即使分化出独立的遗传群体,也未发生过大规模的种群扩张等历史事件,可以作为一个整体进行保护和开发应用。
参考文献
基于线粒体Cyt b基因和D-loop区序列的高邮湖湖鲚(Coilia nasus)遗传多样性分析
[J].
基于线粒体COⅠ和Cyt b基因序列的6种锦鲤(Cyprinus carpio Koi)遗传多样性分析
[J].
四种(鱚)属鱼类线粒体Cyt b基因的序列变异及系统发育研究
[J].测定和分析了4种鱚属(Sillago)鱼类:斑鱚(Sillago aeolus)、亚洲鱚(S. asiatica)、多鳞鱚(S. sihama)和少鳞鱚(S. japonica)线粒体细胞色素b基因序列片段,比较了不同鱚属鱼类种间的序列差异,探讨了彼此间系统发育关系和分类地位。结果显示,4种鱼类平均核苷酸组成为T 29.9%、C 29.2%、A 21.5%、G 19.3%,种间平均净遗传距离在0.157到0.280之间。最大似然法构建的系统树显示,少鳞鱚和亚洲鱚首先聚类、再与多鳞鱚和斑鱚相聚,斑鱚是最晚分化出的鱼类。基于Cyt b基因序列核苷酸分歧速率计算得出4种鱼类发生遗传分化时间在距今785—1400年前的第三纪中新世(Miocene)。
Genetic diversity analysis of Asian clam Corbicula fluminea in the Hongze Lake based on mitochondrial cytochrome b gene
[J].
基于COⅠ和16S rRNA基因片段鉴定厦门海域的仔稚鱼
[J].本文基于线粒体细胞色素C氧化酶亚基 I(COI)基因和16S rRNA基因片段对采集于厦门海域的仔稚鱼样品进行种类鉴定,探究其在仔稚鱼种类鉴定中的适用性。研究共获得64条COI基因序列和74条16S rRNA基因序列,通过序列比对,COI基因将仔稚鱼样品鉴定为26个种类,其中19个种类鉴定到种、6个鉴定到属、1个种类仅鉴定到科;16S rRNA基因将仔稚鱼样品鉴定为29个种类,其中23个种类鉴定到种、6个鉴定到属。COI基因的平均种内遗传距离为0.001 5,平均种间遗传距离为0.197 6,16S rRNA基因的平均种内遗传距离为0.000 3,平均种间遗传距离为0.089 2,COI和16S rRNA基因的平均种间遗传距离都为平均种内遗传距离的10倍以上,两者都可以进行有效的仔稚鱼种类鉴定。在基于COI和16S rRNA基因构建的系统进化树上,所有物种都分别单独聚为一支,同一个种类的不同个体都能聚在同一个分支,这些物种均能得到有效区分。以上结果表明,COI和16S rRNA基因均可以实现仔稚鱼的种类鉴定,两种基因结合使用可以提高仔稚鱼种类鉴定的准确性。
16S rRNA基因和COⅠ基因序列分析在石斑鱼物种鉴定中的应用
[J].对台湾海峡常见的8种石斑鱼进行了16S rRNA基因和COI基因的序列测定,并通过GenBank和BOLD两个数据库进行鱼种鉴定。序列分析表明,COI基因较16S rRNA基因有更大的分辨率;两个基因序列在GenBank中的搜索结果和COI基因序列在两个数据库的搜索结果大部分一致,但仍有部分差异。建议同时使用COI和16S rRNA两种保守基因,进行序列测定,然后在GenBank和BOLD SYSTEMS数据库进行搜索,选择一致的鉴定物种作为鉴定结果。
石首鱼科海洋鱼类DNA条形码的构建
[J].为筛选石首科鱼类物种鉴定的最优DNA条形码,本试验对大黄鱼、小黄鱼、黄姑鱼和棘头梅童鱼4种鱼类71份样本的细胞色素氧化酶亚基I(COI)、线粒体控制区(D-loop)和16S rRNA 3种基因序列进行PCR扩增和测序,分析比较各基因序列比对鉴定能力、种内和种间差异、barcoding gap检验以及序列多态性。结果表明,3种候选DNA条形码序列的种间遗传距离均高于种内遗传距离,符合作为DNA条形码的基本要求。通过遗传差异和barcoding gap检验分析发现,COI和D-loop区适合作为鉴定大黄鱼、小黄鱼、黄姑鱼和棘头梅童鱼4种石首科鱼类的DNA条形码;16S rRNA基因存在一定的缺陷,但是D-loop区和16S rRNA基因序列可能存在比COI通用序列更为有效的DNA条形码区域。考虑引物通用性,推荐COI基因作为最适DNA条形码。本研究结果为海洋鱼类的快速鉴定提供了一定的参考依据。
河蚬(Corbicula fluminea)对氧化石墨烯和全氟辛烷磺酸类物质(PFOS)联合胁迫的生理生化响应
[J].
洪泽湖野生河蚬(Corbicula fluminea)线粒体COⅠ基因序列的遗传多样性分析
[J].
Polymorphism in Corbicula fluminea (Bivalvia:Corbiculoidea) from Hongkong
[J].
Genetic and shell morphological variability of the invasive bivalve Corbicula fluminea (Mǜller,1774) in two Portuguese estuaries
[J].
基于线粒体COⅠ序列的洪泽湖河蚬(Corbicula fluminea)遗传多样性和种群结构分析
[J].
Origin and possible role of males in hermaphroditic androgenetic Corbicula clams
[J].
Statistical method for testing the neutral mutation hypothesis by DNA polymorphism
[J].The relationship between the two estimates of genetic variation at the DNA level, namely the number of segregating sites and the average number of nucleotide differences estimated from pairwise comparison, is investigated. It is found that the correlation between these two estimates is large when the sample size is small, and decreases slowly as the sample size increases. Using the relationship obtained, a statistical method for testing the neutral mutation hypothesis is developed. This method needs only the data of DNA polymorphism, namely the genetic variation within population at the DNA level. A simple method of computer simulation, that was used in order to obtain the distribution of a new statistic developed, is also presented. Applying this statistical method to the five regions of DNA sequences in Drosophila melanogaster, it is found that large insertion/deletion (greater than 100 bp) is deleterious. It is suggested that the natural selection against large insertion/deletion is so weak that a large amount of variation is maintained in a population.
Statistical tests of neutrality of mutations against population growth,hitchhiking and background selection
[J].The main purpose of this article is to present several new statistical tests of neutrality of mutations against a class of alternative models, under which DNA polymorphisms tend to exhibit excesses of rare alleles or young mutations. Another purpose is to study the powers of existing and newly developed tests and to examine the detailed pattern of polymorphisms under population growth, genetic hitchhiking and background selection. It is found that the polymorphic patterns in a DNA sample under logistic population growth and genetic hitchhiking are very similar and that one of the newly developed tests, Fs, is considerably more powerful than existing tests for rejecting the hypothesis of neutrality of mutations. Background selection gives rise to quite different polymorphic patterns than does logistic population growth or genetic hitchhiking, although all of them show excesses of rare alleles or young mutations. We show that Fu and Li's tests are among the most powerful tests against background selection. Implications of these results are discussed.