The research progress on the degradation mechanism of microcystins in water by environmental microbes
-
摘要:
近年来,全球气候变暖和水体富营养化加剧导致蓝藻水华污染严重,而大部分蓝藻水华暴发后产生的微囊藻毒素(Microcystins,MCs)极具危害性,对人类健康和水生生态系统构成威胁。有效控制和去除水体中的MCs已成为当前亟需解决的一项难题。现有研究表明,微生物降解MCs具有高效和环保的治理效益。因此,本文综述了MCs的产生、结构和毒性危害,并着重介绍了国内外学者在微生物降解MCs的酶促降解途径、降解基因和降解酶领域的研究进展;此外,还分析了微生物降解菌在水体生态修复中的潜在应用,并展望了对MCs的非mlr降解途径机制、优化表达MCs降解酶、构建高效双功能降解菌等未来研究方向,旨在为深入研究MCs微生物降解机制并有效改善全球水体中MCs污染现状提供新思路。
Abstract:Over the past few years, the issue of Cyanobacteria bloom pollution has escalated due to the combined impacts of global warming and eutrophication in aquatic systems. This has resulted in the release of toxic microcystins (MCs), posing a significant threat to human health and aquatic ecosystems. Effectively controlling and eliminating MCs from water bodies is a pressing global challenge. Fortunately, prior research has demonstrated that microbial degradation of MCs presents an efficient and environmentally friendly solution. This review explores the production, structure, and toxicity of MCs, with a focus on summarizing global advancements in enzymatic degradation pathways, degradation enzymes, and degradation genes associated with MCs. Additionally, it also analyzes the potential applications of microbial degradation bacteria in the ecological restoration of water bodies. Furthermore, the paper delves into future research directions, including alternative pathways for non-mlr-mediated MCs degradation mechanisms, optimizing the expression of MCs-degrading enzymes, and constructing highly efficient dual-functional degradation bacteria. These perspectives aim to deepen human’s understanding of microbial degradation mechanisms of MCs and provide innovative solutions to address the global challenge of MCs contamination in water bodies.
-
全球人口密度和工农业的快速增长导致大量含有氮、磷营养盐的废水被直接排放到水体中,使水体富营养化程度不断加重。近几年来,全球范围内已有高达63.1%的湖泊水体出现富营养化[1],其中中国的巢湖、太湖、滇池等大型湖泊也处于不同程度的富营养化状态[2-4]。在水体富营养化和气候变暖等因素的驱动下,全球多处淡水湖泊蓝藻水华的发生频率、危害程度和持续时长日益增加[5]。蓝藻水华的暴发会大量消耗水中氧气、污染水质、影响水生动植物的生长发育、破坏细菌的群落结构,以及威胁水生生态系统的可持续发展[6-7]。同时,以蓝藻(Cyanobacteria)为主的产毒藻,包括微囊藻(Microcystis)、鱼腥藻(Anabaena)、浮丝藻(Planktothrix)、颤藻(Oscillatoria)、念珠藻(Nostoc)和束丝藻(Aphanizomenon)等会产生一系列的次生代谢物,包括微囊藻毒素(Microcystins,MCs)、鱼腥藻毒素(Anatoxins)、节球藻毒素(Nodularins)和束丝藻毒素(Aphanizotoxins)等[8],这些毒素对人类和动物均具有不同程度的毒性,其中以MCs最为常见且毒性危害最大,会对人类和动物的多个器官造成严重的毒性危害,甚至引发癌症[9-10]。近年来,研究发现全球水体中的胞外MCs浓度范围已达到0~42 724 μg/L[11],并且受水域温度和光照等环境因素的影响,MCs的时空分布特征明显,其浓度会在夏季达到高峰,从秋季到冬季则呈下降趋势[12-13]。此外,由于MCs污染水体从内陆水域向下游漂流而进入河口和海域,全球各沿海海岸的潟湖和海洋中也检测出MCs,并在当地生物体内逐渐积累[14-16]。迄今为止,全球已发生多起微囊藻毒素中毒和死亡事件[17],对人类社会发展构成严重威胁。因此,如何有效控制和去除水体中的MCs已成为当前亟需解决的一项难题。
MCs是稳定存在于自然水体中的环状七肽肝毒素,治理其的传统方法存在成本高、难以操作且容易造成二次污染等问题。微生物降解被认为是一种可行的治理方法,因为自然水体存在着能以MCs为唯一碳源和氮源的微生物,它们能有效降解水体中的MCs[18-19]。目前,国内外研究人员已对微生物降解菌的降解途径和机制进行了大量研究,并深入探究了微生物降解菌的降解途径和机制,旨在开发新型、升级的治理方法,实现MCs的高效治理和水体生态修复。
本文综述了MCs的产生、结构及毒性危害等基本概况,重点分析了国内外学者在MCs的酶降解途径及相关功能基因领域上的研究进展,旨在增强人们对微生物降解MCs机制的认识。此外,为客观了解采用微生物降解法治理MCs污染水体的有效性,本文分析了近年来各类微生物降解菌在水体生态修复过程中的应用价值,并展望了MCs微生物降解领域的研究工作,为解决全球水体中MCs污染现状及深入研究MCs微生物降解机制提供新思路。
1. MCs的产生、结构及毒性危害
1.1 MCs的产生
MCs是大部分有害蓝藻产生的次级代谢物,在藻细胞裂解或死亡时释放到水环境中[20]。这些产毒藻体内存在一类用于调控MCs产生的合成酶基因簇(mcy),由mcy基因编码的非核糖体多肽合成酶(Nonribosomal peptide synthetase,NRPS)和聚酮合酶(Polyketides,PKS)等功能酶复合物会通过一种非核糖体途径催化合成MCs [21-22]。此外,光照、温度和营养盐等外界环境因子会通过影响MCs合成基因(mcy)的表达水平来直接调控MCs的产生[23-24]。Kaebernick M等[25]研究发现,高强度的光照或红光均能上调铜绿微囊藻(Microcystis aeruginosa)中合成基因mcyB和mcyD的表达水平;Scherer P I等[26]研究发现,当温度在20~30 ℃之间时,提高环境温度能显著上调铜绿微囊藻中mcyB基因的表达;Zhou Y P等[27]的研究表明,氮胁迫能够上调铜绿微囊藻中编码MCs合成的所有基因。因此,自然水体中MCs的产生除受基因的调控表达外,还与外界的环境因子密切相关。
1.2 MCs的结构
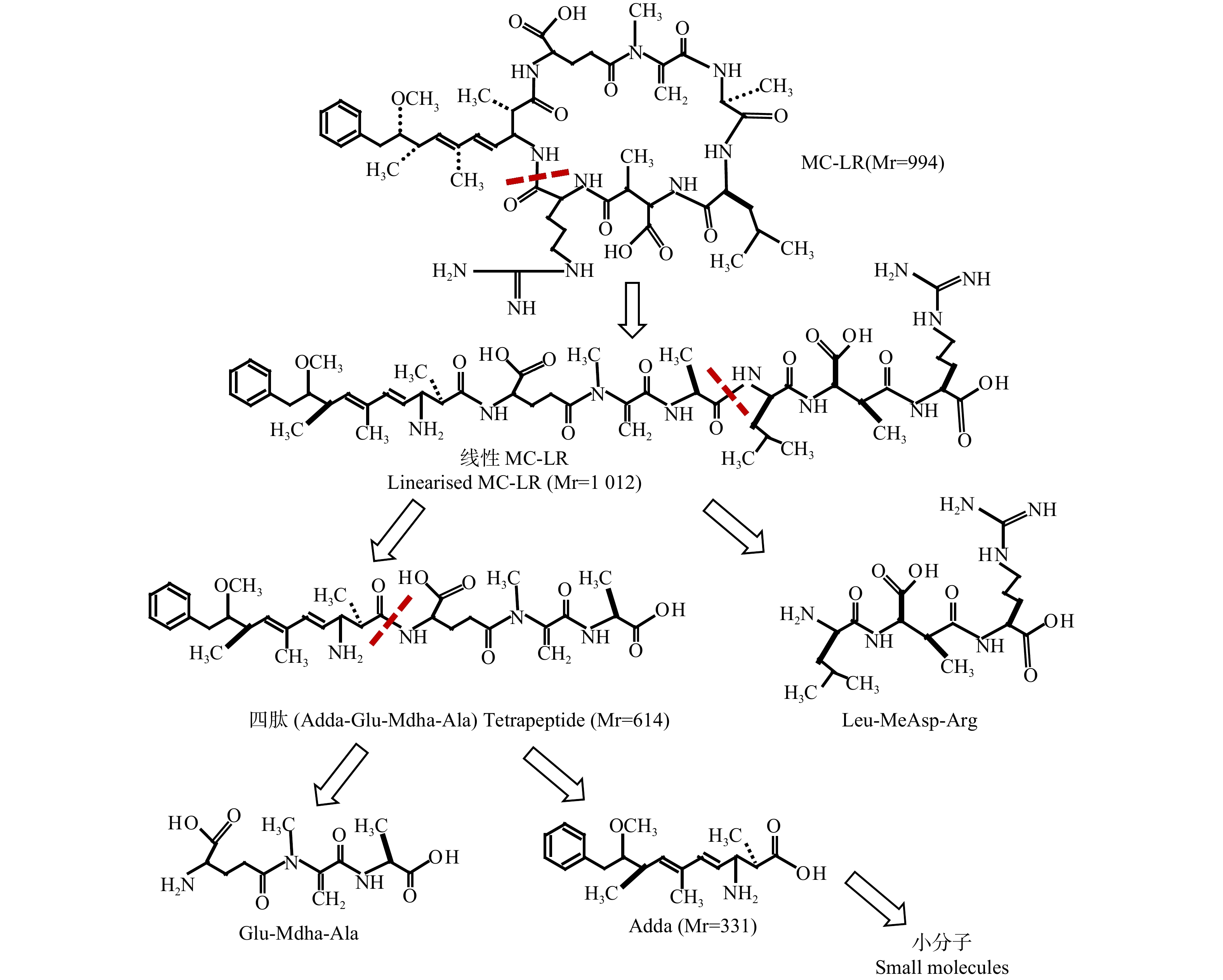
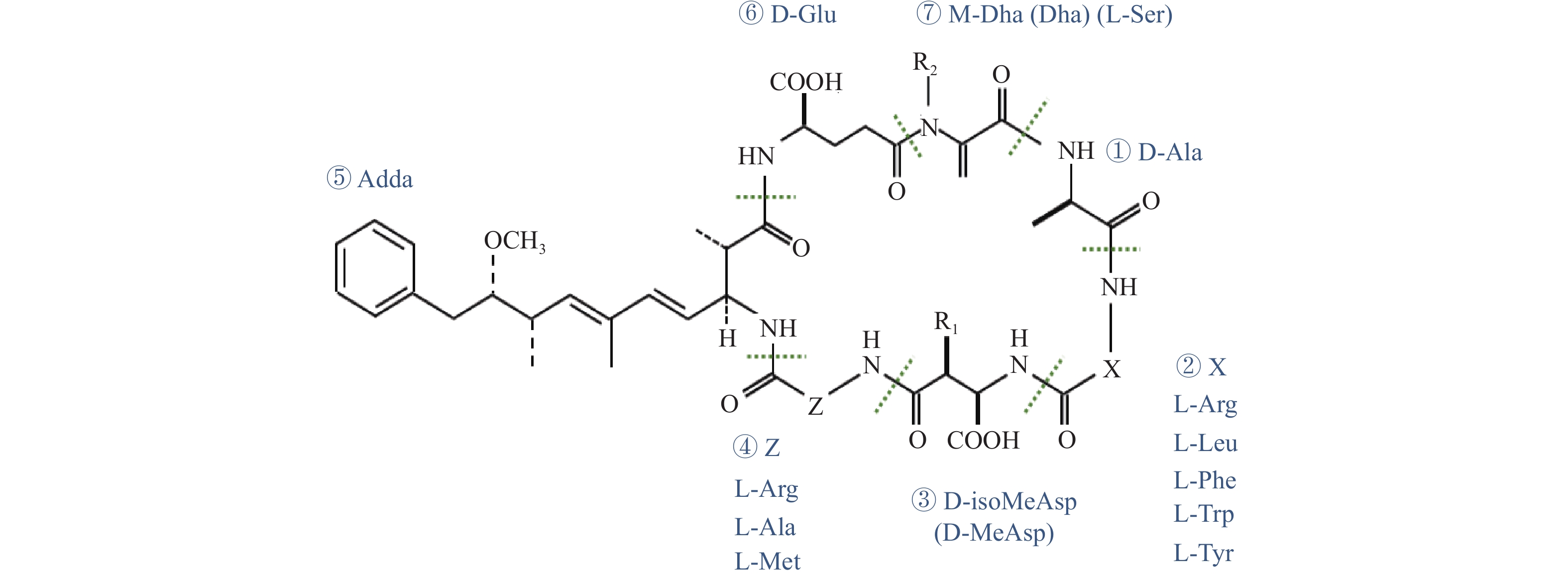
MCs是一类相对分子量在900~1 100 Da之间的环状七肽化合物[28],其结构由肽键连接5个保守氨基酸和2个可变L-氨基酸构成,被定义为环-[D-Ala(1)-L-X(2)-D-MeAsp(3)-L-Z(4)-Adda(5)-D-Glu(6) -Mdha(7)][29]。MCs的结构中含有多个重要基团,包括3号位置的D-MeAsp、7号位置的M-Dha以及5号位置的Adda,其中Adda是表达MCs毒性的关键基团。MCs结构中2、4号位置存在多种类型的氨基酸组合方式以及3、7号位置存在甲基化和去甲基化的差异,因此迄今为止已经发现了不少于279种MCs的变体[30],其中以MC-LR(Leu和Arg)、MC-YR(Tyr和Arg)和MC-RR(Arg和Arg)3种变体最为普遍,且当2、4号位置存在亮氨酸(Leu)和精氨酸(Arg)时便构成了目前最为常见且毒性危害最大的MC-LR变体。MCs的化学结构如图1所示。
1.3 MCs的危害
MCs作为一类细胞内毒素,会抑制胞内特异性蛋白磷酸酶PP1和PP2A活性,影响细胞的正常代谢,造成机体细胞内氧化应激[32],诱导DNA损伤和细胞凋亡[33]。水生动物接触过量的MCs会导致其肝脏坏死、生长发育缓慢、繁殖能力下降甚至死亡,从而破坏水生生态系统的结构和功能[34]。此外,MCs的主要靶器官是肝脏,当人类接触到经MCs污染后的水源或食物后,MCs会借助有机阴离子转运多肽(OATPs)和胆汁酸转运系统主动运输至肝细胞内,导致肝细胞坏死并通过激活体内丝裂原活化蛋白激酶(MAPK)和蛋白激酶B(AKT)等细胞增殖通路引发肝癌[35]。如1996年巴西曾发生了全球首例由MCs污染引发的人类中毒案例,其中52例患者因使用了被MCs污染的透析水,导致急性肝中毒而死亡[36]。中国东南沿海地区成为原发性肝癌高发区的主要原因也与水源被MCs污染有关[37]。此外,MCs还会对肾脏、心脏以及生殖系统等造成巨大的毒性危害,引发心源性并发症、肾脏疾病和心血管疾病等[38-41]。因此,为保障居民生命安全,世界卫生组织和中国卫生部规定饮用水源中MCs的安全阈值应低于1.0 μg/L[42]。
2. 微生物降解MCs的酶类、降解基因及降解途径
酶在微生物降解MCs过程中发挥着重要作用。微生物依赖特殊的胞内酶有效降解MCs,表明MCs的降解与特定酶及酶促机理密切相关。近年来的研究报道过一些微生物对MCs的酶促途径和分子机制以及降解产物、降解酶和编码降解酶的功能基因。
2.1 微生物降解MCs的酶类
2.1.1 MCs降解酶MlrA
MlrA是微生物降解MCs过程中的关键蛋白酶,能切割MC-LR环肽中的Adda-Arg键,使其开环转化为线性MC-LR,从而显著降低其毒性危害。MlrA酶由336个氨基酸残基构成,含有活性位点H260、AIH263、NE265[43-44]。MlrA酶的N端存在着由26个氨基酸组成的信号肽序列,但不同菌株存在细微差异,如根瘤菌(Rhizobium sp. TH)中MlrA酶的N端信号肽只含有23个氨基酸[45],这种信号肽可以帮助MlrA酶进行跨膜定位,在MCs的降解过程中发挥着重要作用;其序列下游存在高度保守的Abi结构域,能实现MlrA酶的催化[46]。Xu Q Q等[47]利用同源建模和定点突变技术验证了MlrA酶上的其他活性位点,包括Glu172、Trp176、His205、Trp201。潘禹等[48]通过构建MlrA酶的结构模型发现,MlrA结构中的Abi结构域会形成反平行式的螺旋构型,在其他4个螺旋的围绕下共同构成巨大的锥形催化空腔,使底物和溶剂从反应腔底部的周质空间进入酶分子内部,通过与酶上的活性位点的结合作用来实现底物的催化降解,如MlrA酶可以通过传统氢键、π-π堆积以及范德华力等与MC-LR进行相互作用,将环状MC-LR降解为线性MC-LR[49]。
2.1.2 MCs降解酶MlrB
MlrB酶是微生物降解MCs过程中的第二个关键作用酶,它可以通过断开线性MC-LR中的Ala-Leu键,使其降解为四肽,因此MlrB酶也被称为线性化微囊藻毒素酶(Linearized-microcystinase)。通过Blastp分析发现,MlrB酶的蛋白质序列与青霉素识别酶家族成员和β-内酰胺酶家族成员均具有较高的序列相似性,并且还含有一个高度保守的Ser-Xaa-Xaa-Lys活性位点序列[43,50]。Dziga D等[51]根据该序列设计了一个突变实验,将MlrB酶的S77和K80分别突变为丙氨酸(Ala),结果发现酶活性明显丧失,说明Ser77和Lys80这2种氨基酸残基对MlrB酶的催化功能具有重要作用。Wei J等[52]通过同源建模和分子对接实验分析了MlrB酶的结构和结合方式,发现MlrB酶N末端和C末端区域主要定位于细胞表面,由一个跨膜结构域S1通过重入螺旋进行连接,且跨膜螺旋被预测采用了拓扑结构,构成MlrB酶的氨基酸残基会通过形成一个活性口袋来帮助线性MC-LR与活性位点进行结合,完成线性MC-LR的催化降解。
2.1.3 MCs降解酶MlrC
MlrC酶是存在于MCs微生物降解途径中的一类特殊降解酶,除降解四肽外,还可以直接将线性MC-LR降解为Adda,对于无法表达mlrB基因的微生物降解菌具有重要意义。MlrC酶是一种金属蛋白酶,具有507个氨基酸残基[43],其酶结构主要由DUF1485和MlrC-C两类结构域组成,但不含氨基酸信号肽和跨膜区。其中,DUF1485结构域家族(约300个残基)是由变形菌门中功能未知的假想细菌蛋白质组成,而MlrC-C结构域家族(约200个残基)是发挥酶催化功能的一个重要区域,主要位于MC-LR降解的细菌基因簇产物的C末端[50,53]。Dziga D等[51]通过突变实验验证了MlrC酶中的3个关键性残基(Asp167、His169、His191)。为了深入研究MlrC酶的结构特征及作用机制,Singh D P等[54]利用I-TASSER软件预测了MlrC酶的三级结构,同时设计的对接实验发现MC-LR酶主要通过氢键与MlrC酶的氨基酸残基Thr399和Asp400进行结合。Wang R P等[55]以PDB数据库中同源性较高的3IUU蛋白为建模模板,分析了MlrC酶的三级结构,并通过突变实验验证了6个活性位点(Glu39、His133、Asp167、His169、His191和Asp332),而这些活性位点的空间位置可能构成了金属蛋白酶的多个催化三联体,并且由活性位点形成的酶活性口袋可以通过结合金属离子共同催化线性MCs的降解。
2.2 微生物降解MCs的降解基因
近年来,微生物降解MCs机制的研究受到广泛关注。最初,Bourne D G等[43]鉴定出ACM-3962菌的4段降解基因,分别为mlrA、mlrB、mlrC、mlrD,其中由基因mlrA、mlrB和mlrC编码的3种降解酶MlrA、MlrB和MlrC可将MC-LR降解为氨基酸和小分子肽,mlrD编码的MlrD酶被推测为一种寡肽转运蛋白酶,负责MC-LR及其降解产物的转运。除mlrA~mlrD 4个基因外,Okano K等[56]和Jin H Y等[57]先后报道了鞘脂单胞菌(Sphingopyxis sp. C-1)和鞘氨醇单胞菌(Sphingosinicella sp. B-9)的全基因组测序,揭示了其他降解基因如mlrE和mlrF可编码修饰二肽酶和D-氨基酰化酶;B-9菌的MlrE酶可能参与氨基酸类化合物的催化降解[58]。利用全基因组测序的方法可以更好地鉴定和表征MCs微生物降解菌中存在的mlr降解基因,对于描述MCs降解菌的降解途径及分子机理具有重要意义。
mlrA降解基因是MCs降解途径中的关键性基因。相关研究表明,缺陷短波单胞菌(Brevundimonas diminuta Q3)[18]、鞘脂单胞菌USTB-05[59]、鞘氨醇单胞菌MD-1和Y2菌[60]等均能扩增得到mlrA基因,且与鞘氨醇单胞菌ACM-3962的mlrA基因有较高的相似性。表明这些菌株与鞘氨醇单胞菌ACM-3962可能具有相同的降解机制。但并非所有MCs降解菌均含有该基因,如Krishnan A等[61]从伊利湖中获得的6株MCs降解菌未被检测出mlrA基因;Huang F Y等[62]分离得到的厌氧MCs降解菌Enterobacter sp. YF3菌株中也未被检测出mlrA、mlrB、mlrC、mlrD基因。说明这些菌株可能存在其他新型的MCs降解途径,但也存在检测时引物和模板不匹配造成的假阴性。除mlrA降解基因外,Qin L等[63]通过对mlr基因簇进行系统发育分析,发现mlr基因簇的4个基因(mlrA~mlrD)具有相同的起源和进化史。这对于研究新型MCs降解菌的降解机理具有重要意义。
此外,为了验证mlrA、mlrB、mlrC基因的活性和功能,Wang R P等[64]构建了Novosphingobium sp.THN1菌的mlrA基因重组菌pET-29a-mlrA-BL21,发现重组MlrA酶可以切割Adda-Arg之间的肽键,使MC-RR水解为线性MC-RR;Shimizu K等[50]通过构建鞘脂单胞菌C-1的mlrB和mlrC基因重组菌,发现重组MlrB酶能将MCs降解为四肽,而重组MlrC酶能将四肽降解为Adda或直接将线性MC-LR降解为Adda。利用异源表达的方法可以有效地验证基因的活性功能,阐明MCs降解菌的降解机理。
2.3 微生物降解MCs的降解途径
2.3.1 MCs的酶促途径
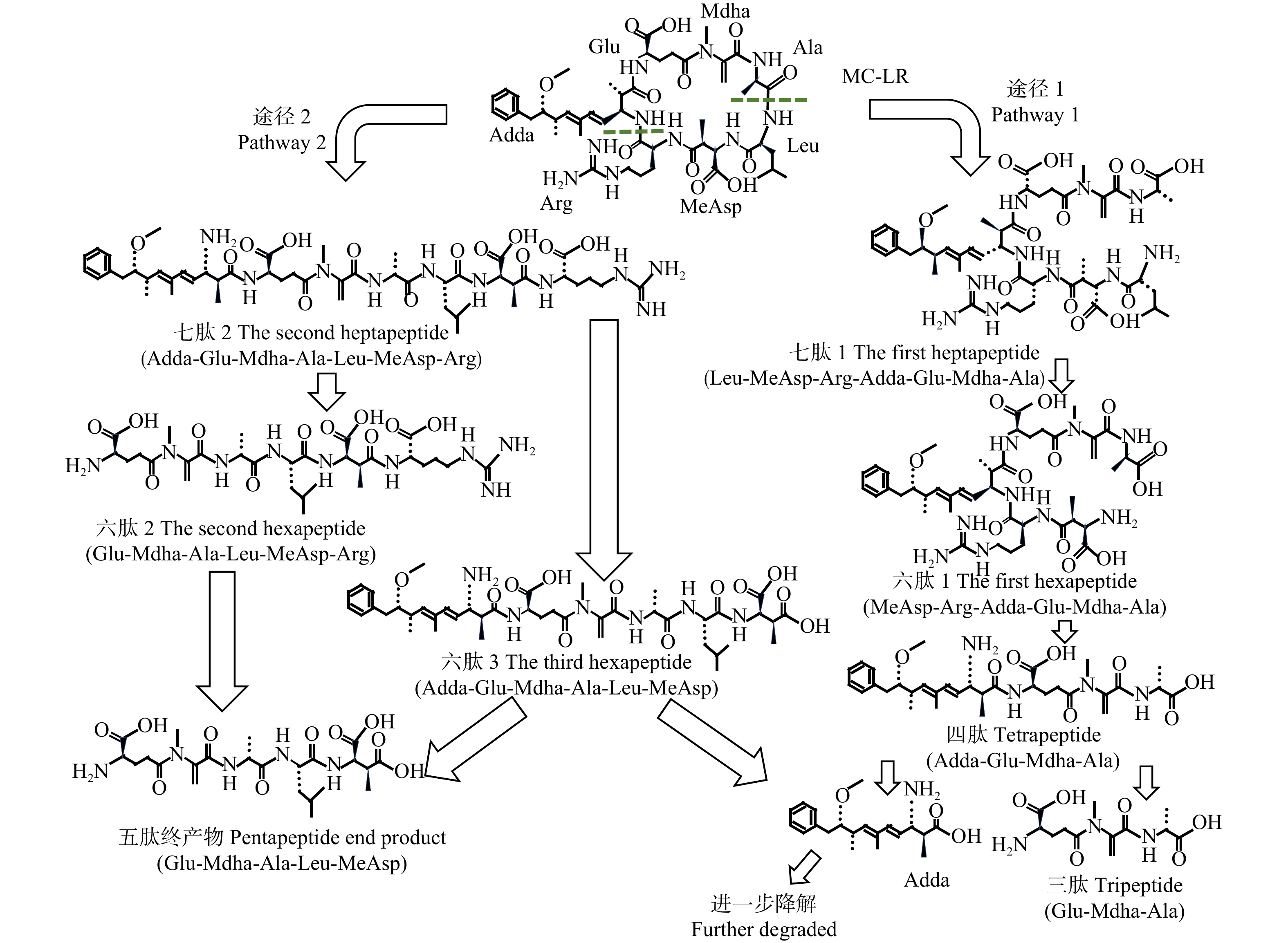
对于MCs的酶促途径,Bourne D G等[43,65]首次提出了鞘氨醇单胞菌ACM-3962对MC-LR的酶促途径是由mlr基因簇编码的3种胞内水解酶(MlrA、MlrB和MlrC)和2种中间产物(线性MC-LR和四肽)参与,MlrA酶先水解MC-LR中Adda-Arg之间的肽键,使其线性化,产生毒性降低约160倍的线性MC-LR;然后MlrB酶将线性MC-LR中Ala-Leu之间的肽键断开,生成四肽产物;最后,MlrC酶将四肽中Adda-Glu之间的肽键断开,生成更小的肽片段和氨基酸。但后续研究发现,MlrC酶也可以直接将线性MC-LR水解为Adda[50-51],表明存在一种可替代途径,可以不经过MlrB酶而直接降解线性MC-LR,这对于无法表达MlrB酶的降解菌株而言具有重要意义。不同菌株对于MCs的降解产物存在差异,如Zhang J等[66]发现鞘脂单胞菌a7可同时降解MC-LR和Adda,产生5种降解产物,这是首次报道单菌株同时降解MC-LR和Adda的一条降解途径(图2)。Ding Q等[67]在研究过程中也检测出了8种新的降解产物,包括3种三肽(Adda-Glu-Mdha、Glu-Mdha-Ala和Leu-MeAsp-Arg)、3种二肽(Glu-Mdha、Mdha-Ala和MeAsp-Arg)以及2种氨基酸(Leu和Arg),其中Mdha-Ala、MeAsp-Arg和Leu为首次报道。这些新降解产物的产生表明可能存在其他新型降解酶参与了MCs的降解,同时也存在Mlr降解酶表达多种降解功能的可能性。
除MC-LR外,国内外对于MC-RR和MC-YR的酶促途径也开展了大量的研究工作。其中,Yan H等[59]和Wang H S等[68]相继利用了鞘脂单胞菌USTB-05对MC-RR的酶促途径进行了详细研究,结果表明MC-RR的降解途径与上述报道的MC-LR途径十分相似。而对于MC-YR的降解途径,Xu H M等[69]研究表明MC-YR的前三步降解过程也与MC-LR、MC-RR类似,只是切割位点存在差异。此外,Zhu X Y等[45]研究了一株根瘤菌TH对MC-LR的降解途径,结果表明TH菌株可能利用了与ACM-3962菌株相同的mlr降解机制对MC-LR进行降解。
此外,研究发现,厌氧条件下富营养化湖泊沉积物中的微生物群落也能有效地降解水体中的MCs[70],例如Bao Z Y等[71]研究发现厌氧微生物降解菌ALA-1对MC-LR的降解途径与大部分好氧菌相似,产生了线性MC-LR、四肽和Adda产物;欧阳潦[72]利用从滇池中分离出的一株MCs厌氧降解菌(Acidaminobacter sp.CJ5)探究MC-LR的厌氧降解途径,结果发现MC-LR在胞内酶的作用下转化为线性MC-LR和Adda,但其产生的线性MC-LR与好氧途径中的线性MC-LR均不相同,初步推测可能存在一种新型MC-LR降解途径;Ding Q等[73]以太湖中的微生物群落为研究对象,提出了2种较为完整的MC-LR厌氧微生物降解途径(图3)。以上研究表明,自然环境中的厌氧微生物降解菌也能通过特殊的降解途径来有效地降解MCs,但该领域的研究报道仍十分有限,对于MCs厌氧降解菌的降解机理还需要进一步的研究。
2.3.2 MCs的降解基因调控途径
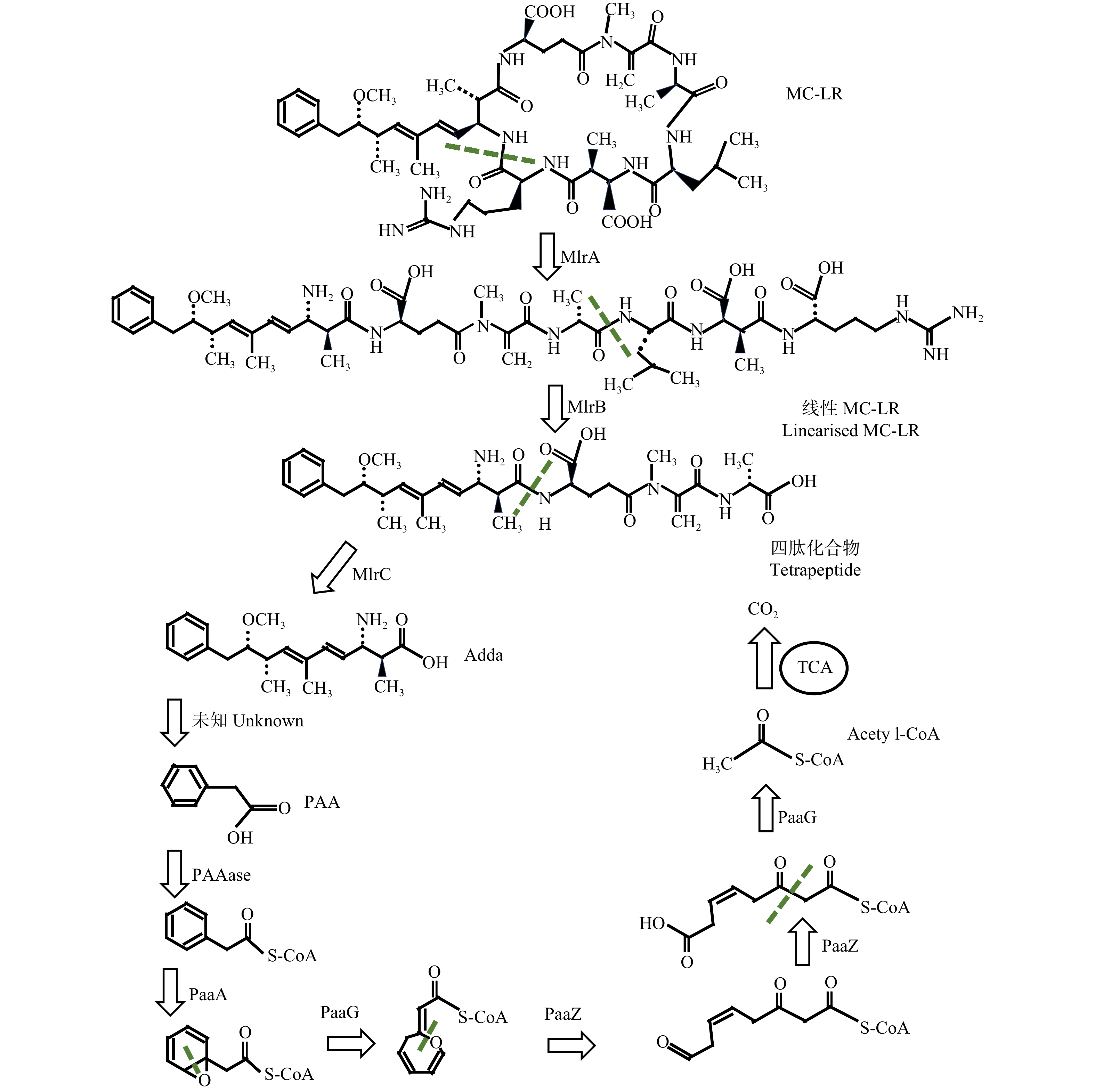
迄今为止,已报道的大部分MCs的微生物降解菌均是依赖于mlr基因簇编码的Mlr酶(简称mlr途径)进行降解。据统计,MCs高效降解菌如鞘氨醇单胞菌属(Sphingomonas)、嗜麦芽窄食单胞菌(Stenotrophomonas maltophilia)、芽孢杆菌属(Bacillus)、不动杆菌属(Acinetobacter)、金黄杆菌属(Chryseobacterium)、根瘤菌属(Rhizobium)和博德特氏菌属(Bordetella)等均能被检测出mlr降解基因[30]。这表明mlr基因簇在MCs的微生物降解途径中可能发挥着关键性作用。后续研究发现,这些依赖mlr途径的降解菌株会受到一种由酰基高丝氨酸内酯(Acyl homoserine lactone,AHL)介导的群体感应(Quorum sensing,QS)系统的调节,它通过转录激活mlr基因的表达,尤其是mlrA基因的表达来正向调节MCs的降解,并且通过基因敲除和互补技术发现,LuxI/LuxR型QS系统(novI1/novR1和novI2/novR2)可以有效地促进MCs降解菌降解MCs[74]。因此,后续若能合理操纵QS系统的调控机制来提高mlr基因的表达活性,将能达到MCs的高效降解。然而,除mlr基因外,近年来也有研究发现了许多不依赖mlr基因簇的MCs降解菌,如敏捷食酸菌(Acidovorax facilis LEw-2)[61]、芽孢杆菌(Bacillus sp.LEw-2010)[61]、耐硼赖氨酸芽孢杆菌(Lysinibacillus boronitolerans CQ5)[75]、微嗜酸寡养单胞菌(Stenotrophomonas acidaminiphila MC-LTH2)[76]等,这些降解菌株未被检测出mlr降解基因,但同样能有效降解水体中的MCs,这表明自然环境中可能存在着其他非mlr降解途径,但由于目前的研究报道十分有限,是否存在新型MCs降解基因及降解酶仍有待探究。但Yang F等[77]发现Adda会通过PAA代谢途径逐步降解为乙酰辅酶A(Acetyl-CoA),而乙酰辅酶A最终通过三羧酸循环(Tricarboxylic acid cycle,TCA)被完全氧化为二氧化碳。这是首次提出的一条由mlr基因和paa基因共同调控的酶促降解途径(图4)。此外,利用QS系统可以正向调节PAA降解途径中的第一步和最后一步反应,从而推动MC-LR向无毒产物二氧化碳的转化[78],实现MCs的无害降解。
3. 微生物降解菌在水体生态修复中的潜在应用
自然环境中微生物菌种资源丰富,作为生态系统中的分解者,它们在降解水体污染物质、改善水质和修复水环境中发挥着重要作用。其中,MCs微生物降解菌可有效降解水体生态系统中的MCs,在MCs污染水体的生态修复中有着潜在的应用价值。
目前自然界中分离出大量能够高效降解MCs的细菌。如Zhang J等[66]从太湖中分离出的鞘脂单胞菌a7能以3.33 mg/L·h−1的降解速率将MC-LR和代谢产物完全降解;Idroos E S等[79]从斯里兰卡贝拉湖分离出的嗜麦芽窄食单胞菌(Stenotrophomonas maltophilia 4B4)能100%降解水体中的MCs;Yang F等[80]从太湖中分离出的降解菌群YFMCD4能以0.5 μg/mL·h−1的降解速率将MC-LR完全去除。越来越多的高效纯菌株被分离出,若将这些纯菌株进行混合研究并发挥其协同降解作用,有望研发出更加高效的混合降解菌群,以治理水体的MCs污染。此外,环境中存在多种双功能型降解菌,其能同时降解水体中的有害藻类和MCs,如Kang Y H等[81]筛选出的铜绿假单胞菌(Pseudomonas aeruginosa UCBPP-PA14)和恶臭假单胞菌(Pseudomonas putida KCCM10464)能同时实现铜绿微囊藻和MCs的双重降解;此外还有不动杆菌(Acinetobacter CMDB-2)[82]、链霉菌(Streptomyces amritsarensis HG-16)[83]、根瘤菌AQ_MP[84]、芽孢杆菌AK3[85]等。这些双功能降解菌不仅能从根源上抑制蓝藻水华的发生,还能有效地除去藻属释放的MCs,对于控制和修复由蓝藻水华和MCs引发的水体污染具有重要的应用价值。
考虑到外界因素(如温度、pH、营养物质和细菌细胞密度等)可能对高效降解菌的降解能力产生抑制作用,有学者就此提出了一些新型的MCs降解菌应用方案。例如,Ren G F等[86]研究发现固定在活性炭纤维海藻酸钠的鞘脂单胞菌YF1对MC-RR和污染物质的去除效果要明显优于自由悬浮的YF1,并且在极端温度和pH的不利环境下也能保持较高的降解性;Boonbangkeng D等[85]以漂浮多孔玻璃颗粒(FPGPs)为固定化材料,将芽孢杆菌AK3进行固定后,发现与自由悬浮的细胞相比,AK3菌同样表现出更优的除藻效果和更高的MC-RR降解效率。近年来,微生物固定化技术凭借着微生物密度大、降解效率高、稳定性好等优点逐渐成为当前的研究热点。而生物强化作为一种新兴的技术,可以通过向系统中添加特定功能的优势微生物来提高系统对污染物质的去除效率[87],现已被广泛研究和应用于生态环境修复中。例如,万翔[88]通过向人工湿地系统添加MCs高效复合降解菌进行生物强化后,发现强化后的湿地系统对MCs的降解速率明显得到提高,并且还能够通过缩短其半衰期来紧急处理蓝藻水华和藻毒素造成的突发情况;Wang R等[89]研究发现经过生物强化后的人工湿地系统可以显著提高其对MC-LR的降解效率,在12 h内即可将质量浓度为16.7 μg/L的MC-LR降低至1.0 μg/L,并且可以消除微生物降解的延滞期;Kumar P等[90]将天然降解菌株与MCs降解菌共培养,该方法显著增强了过滤器对水中MC-LR的降解能力。这些研究均证实了生物强化技术能够有效地提高污水处理系统对污染物质的降解效能,并且利用群体感应(QS)系统能够帮助生物强化系统实现外源功能菌的成功定植及优化系统中微生物的群落结构[91],这对于推动水体生态修复工作的历史进程具有重要意义。
4. 展望
蓝藻水华及其产生的MCs已经对水生生态系统和人类健康造成了严重威胁。当前,综合考虑经济成本和环境保护等因素,相比于传统的物化处理工艺,微生物降解被认为是一种较为优质的MCs去除方法。已有的研究表明,MCs降解菌通过多种酶促降解途径和分子机制,能够将有毒的MCs逐步降解为无毒产物。同时,将MCs微生物高效降解菌投入生物系统中进行生物强化,也能够极大地提高系统对MCs的降解效能。迄今为止,国内外在MCs的微生物降解领域也已取得了重大进展,众多学者已从酶学和分子水平上研究分析了MCs的微生物降解途径和降解机制,这为构建高效降解MCs的基因工程菌和工业生产酶制剂提供了理论基础。结合现有的研究结果,未来还需要在以下几个方面进行深入研究:
1)阐明MCs的非mlr降解途径及降解机制。目前MCs微生物降解的研究主要集中于mlr降解途径,但近年来已有的研究发现,MCs的厌氧降解菌及部分好氧降解菌未被检测出mlr降解基因,表明这些降解菌很可能通过非mlr降解途径降解MCs,而其降解过程中所涉及的降解基因及降解酶还尚不清楚,因此还需通过分离出更多的MCs微生物降解菌,通过详细的基因分析和酶学分析来进一步来阐明MCs的非mlr降解途径及降解机制。
2)MCs降解酶的表达优化。酶是微生物降解MCs过程中的关键物质,研究其结构和功能尤为重要,因此可以通过现代化生物信息学分析技术及分子模拟技术来精确解析酶的晶体结构,并利用分子对接技术,深入探究酶与底物分子的催化机理,而后采用基因工程技术对酶分子进行定向改造,以提高酶的稳定性和降解活性,从而研发出一种高稳定性、高效率的MCs降解酶。
3)微生物降解MCs的群落结构对降解效率的影响还需要进一步深入研究和探索。利用高通量测序技术对微生物降解MCs的群落结构进行深入解析,可以通过分析水环境中的微生物样品,捕捉到参与MCs的微生物种类及其丰度的变化,对比不同水体样品,了解微生物群落结构与降解效率之间的关系。
-
-
[1] Wang S L, Li J S, Zhang B, et al. Trophic state assessment of global inland waters using a MODIS-derived Forel-Ule index[J]. Remote Sensing of Environment, 2018, 217: 444 − 460. DOI: 10.1016/j.rse.2018.08.026
[2] 朱金杰,邹楠,钟寰,等. 富营养化巢湖沉积物溶解性有机质光谱时空分布特征及其环境意义[J]. 环境科学学报,2020,40(7):2528 − 2538. [3] 杨柳燕,杨欣妍,任丽曼,等. 太湖蓝藻水华暴发机制与控制对策[J]. 湖泊科学,2019,31(1):18 − 27. DOI: 10.18307/2019.0102 [4] 袁海英,侯磊,梁启斌,等. 滇池近岸水体微塑料污染与富营养化的相关性[J]. 环境科学,2021,42(7):3166 − 3175. [5] Kaloudis T, Hiskia A, Triantis T M. Cyanotoxins in bloom: ever-increasing occurrence and global distribution of freshwater cyanotoxins from planktic and benthic cyanobacteria[J]. Toxins, 2022, 14(4): 264. DOI: 10.3390/toxins14040264
[6] 王端明,陈昌文. 蓝藻水华产生原因、危害及防控措施[J]. 广东化工,2021,48(10):151 − 153. DOI: 10.3969/j.issn.1007-1865.2021.10.056 [7] 彭宇科,路俊玲,陈慧萍,等. 蓝藻水华形成过程对氮磷转化功能细菌群的影响[J]. 环境科学,2018,39(11):4938 − 4945. [8] 罗丛强,王素钦,左俊,等. 鱼腥藻毒素的生态毒性研究及展望[J]. 生态毒理学报,2022,17(5):217 − 225. DOI: 10.7524/AJE.1673-5897.20211014002 [9] Ma Y, Liu H H, Du X D, et al. Advances in the toxicology research of microcystins based on omics approaches[J]. Environment International, 2021, 154: 106661. DOI: 10.1016/j.envint.2021.106661
[10] Zhang W Z, Liu J, Xiao Y X, et al. The impact of Cyanobacteria blooms on the aquatic environment and human health[J]. Toxins, 2022, 14(10): 658. DOI: 10.3390/toxins14100658
[11] 童银栋,黄兰兰,杨宁,等. 全球水体微囊藻毒素分布特征及其潜在环境风险分析[J]. 生态环境学报,2023,32(1):129 − 138. [12] 王永强,国晓春,卢少勇,等. 高原富营养化湖泊杞麓湖微囊藻毒素分布特征、相关因子及其健康风险分析[J]. 湖泊科学,2024,36(1):52 − 63. DOI: 10.18307/2024.0113 [13] Onofrio M D, Egerton T A, Reece K S, et al. Spatiotemporal distribution of phycotoxins and their co-occurrence within nearshore waters[J]. Harmful Algae, 2021, 103: 101993. DOI: 10.1016/j.hal.2021.101993
[14] Edwards M L, Schaefer A M, McFarland M, et al. Detection of numerous phycotoxins in young bull sharks (Carcharhinus leucas) collected from an estuary of national significance[J]. Science of the Total Environment, 2023, 857: 159602. DOI: 10.1016/j.scitotenv.2022.159602
[15] Laureano-Rosario A E, McFarland M, Bradshaw D J, et al. Dynamics of microcystins and saxitoxin in the Indian River Lagoon, Florida[J]. Harmful Algae, 2021, 103: 102012. DOI: 10.1016/j.hal.2021.102012
[16] Bukaveckas P A, Lesutienė J, Gasiūnaitė Z R, et al. Microcystin in aquatic food webs of the Baltic and Chesapeake Bay regions[J]. Estuarine, Coastal and Shelf Science, 2017, 191: 50 − 59. DOI: 10.1016/j.ecss.2017.04.016
[17] Svirčev Z, Lalić D, Bojadžija Savić G, et al. Global geographical and historical overview of cyanotoxin distribution and cyanobacterial poisonings[J]. Archives of Toxicology, 2019, 93(9): 2429 − 2481. DOI: 10.1007/s00204-019-02524-4
[18] 游狄杰,陈晓国,向荟圯,等. 微囊藻毒素降解菌Paucibacter sp. CH菌的分离鉴定及其降解特性[J]. 环境科学,2014,35(1):313 − 318. [19] 谷青,赵琪,史全良. 一株降解微囊藻毒素的缺陷短波单胞菌的分离鉴定及降解特性研究[J]. 环境科学学报,2017,37(1):201 − 206. [20] Preece E P, Hardy F J, Moore B C, et al. A review of microcystin detections in estuarine and marine waters: environmental implications and human health risk[J]. Harmful Algae, 2017, 61: 31 − 45. DOI: 10.1016/j.hal.2016.11.006
[21] 袁志勇,朱红惠,霍光华,等. 微囊藻毒素生物合成及其检测的分子生物学研究进展[J]. 生物技术进展,2012,2(5):328 − 334. DOI: 10.3969/j.issn.2095-2341.2012.05.04 [22] Zhou C C, Chen H, Zhao H P, et al. Microcystin biosynthesis and toxic effects[J]. Algal Research, 2021, 55: 102277. DOI: 10.1016/j.algal.2021.102277
[23] 付保荣,鲁男,苗斌,等. 环境因子对铜绿微囊藻生长和产毒的影响[J]. 辽宁大学学报(自然科学版),2015,42(1):85 − 90. DOI: 10.3969/j.issn.1000-5846.2015.01.015 [24] 张虎. 氮磷浓度比对铜绿微囊藻生长及产毒影响研究[D]. 合肥:合肥工业大学,2020. [25] Kaebernick M, Neilan B A, Borner T, et al. Light and the transcriptional response of the microcystin biosynthesis gene cluster[J]. Applied and Environmental Microbiology, 2000, 66(8): 3387 − 3392. DOI: 10.1128/AEM.66.8.3387-3392.2000
[26] Scherer P I, Raeder U, Geist J, et al. Influence of temperature, mixing, and addition of microcystin-LR on microcystin gene expression in Microcystis aeruginosa[J]. MicrobiologyOpen, 2017, 6(1): e00393. DOI: 10.1002/mbo3.393
[27] Zhou Y P, Li X, Xia Q Q, et al. Transcriptomic survey on the microcystins production and growth of Microcystis aeruginosa under nitrogen starvation[J]. Science of the Total Environment, 2020, 700: 134501. DOI: 10.1016/j.scitotenv.2019.134501
[28] Buratti F M, Manganelli M, Vichi S, et al. Cyanotoxins: producing organisms, occurrence, toxicity, mechanism of action and human health toxicological risk evaluation[J]. Archives of Toxicology, 2017, 91(3): 1049 − 1130. DOI: 10.1007/s00204-016-1913-6
[29] Du X D, Liu H H, Yuan L, et al. The diversity of cyanobacterial toxins on structural characterization, distribution and identification: a systematic review[J]. Toxins, 2019, 11(9): 530. DOI: 10.3390/toxins11090530
[30] Bouaïcha N, Miles C O, Beach D G, et al. Structural diversity, characterization and toxicology of microcystins[J]. Toxins, 2019, 11(12): 714. DOI: 10.3390/toxins11120714
[31] Massey I Y, Yang F. A mini review on microcystins and bacterial degradation[J]. Toxins, 2020, 12(4): 268. DOI: 10.3390/toxins12040268
[32] 黄兰英,张达娟,张树林,等. 微囊藻毒素对凡纳滨对虾肝胰腺氧化损伤研究[J]. 水产科学,2022,41(6):1017 − 1022. [33] 国晓春,卢少勇,谢平,等. 微囊藻毒素的环境暴露、毒性和毒性作用机制研究进展[J]. 生态毒理学报,2016,11(3):61 − 71. [34] Onyango D M, Orina P S, Ramkat R C, et al. Review of current state of knowledge of microcystin and its impacts on fish in Lake Victoria[J]. Lakes & Reservoirs: Science, Policy and Management for Sustainable Use, 2020, 25(3): 350-361.
[35] Shi L J, Du X D, Liu H H, et al. Update on the adverse effects of microcystins on the liver[J]. Environmental Research, 2021, 195: 110890. DOI: 10.1016/j.envres.2021.110890
[36] Yuan M C, Carmichael W W, Hilborn E D. Microcystin analysis in human sera and liver from human fatalities in Caruaru, Brazil 1996[J]. Toxicon, 2006, 48(6): 627 − 640. DOI: 10.1016/j.toxicon.2006.07.031
[37] 谢平. 微囊藻毒素对人类健康影响相关研究的回顾[J]. 湖泊科学,2009,21(5):603 − 613. DOI: 10.3321/j.issn:1003-5427.2009.05.001 [38] Alosman M, Cao L H, Massey I Y, et al. The lethal effects and determinants of microcystin-LR on heart: a mini review[J]. Toxin Reviews, 2021, 40(4): 517 − 526. DOI: 10.1080/15569543.2019.1711417
[39] Zhang S Y, Du X D, Liu H H, et al. The latest advances in the reproductive toxicity of microcystin-LR[J]. Environmental Research, 2021, 192: 110254. DOI: 10.1016/j.envres.2020.110254
[40] Cao L H, Massey I Y, Feng H, et al. A review of cardiovascular toxicity of microcystins[J]. Toxins, 2019, 11(9): 507. DOI: 10.3390/toxins11090507
[41] 梁小芳,杨越,徐帅帅,等. 微囊藻毒素-LR长期低剂量暴露诱导小鼠肾脏结构和功能损伤:基于激活PI3K/AKT信号通路[J]. 南方医科大学学报,2022,42(10):1486 − 1494. DOI: 10.12122/j.issn.1673-4254.2022.10.07 [42] 娄厚顺. 微囊藻毒素水处理方法研究初探[J]. 资源节约与环保,2018(6):99. [43] Bourne D G, Riddles P, Jones G J, et al. Characterisation of a gene cluster involved in bacterial degradation of the cyanobacterial toxin microcystin LR[J]. Environmental Toxicology, 2001, 16(6): 523 − 534. DOI: 10.1002/tox.10013
[44] Dziga D, Wladyka B, Zielińska G, et al. Heterologous expression and characterisation of microcystinase[J]. Toxicon, 2012, 59(5): 578 − 586. DOI: 10.1016/j.toxicon.2012.01.001
[45] Zhu X Y, Shen Y T, Chen X G, et al. Biodegradation mechanism of microcystin-LR by a novel isolate of Rhizobium sp. TH and the evolutionary origin of the mlrA gene[J]. International Biodeterioration & Biodegradation, 2016, 115: 17 − 25.
[46] Sun H H, Wang H S, Zhan H F, et al. Bioinformatic analyses and enzymatic properties of microcystinase[J]. Algal Research, 2021, 55: 102244. DOI: 10.1016/j.algal.2021.102244
[47] Xu Q Q, Fan J H, Yan H, et al. Structural basis of microcystinase activity for biodegrading microcystin-LR[J]. Chemosphere, 2019, 236: 124281. DOI: 10.1016/j.chemosphere.2019.07.012
[48] 潘禹,王华生,詹鸿峰,等. 微囊藻毒素降解酶MlrA的结构功能分析[J]. 化工学报,2021,72(3):1643 − 1653. [49] Cai D P, Wei J, Huang F Y, et al. The detoxification activities and mechanisms of microcystinase towards MC-LR[J]. Ecotoxicology and Environmental Safety, 2022, 236: 113436. DOI: 10.1016/j.ecoenv.2022.113436
[50] Shimizu K, Maseda H, Okano K, et al. Enzymatic pathway for biodegrading microcystin LR in Sphingopyxis sp. C-1[J]. Journal of Bioscience and Bioengineering, 2012, 114(6): 630 − 634. DOI: 10.1016/j.jbiosc.2012.07.004
[51] Dziga D, Zielinska G, Wladyka B, et al. Characterization of enzymatic activity of MlrB and MlrC proteins involved in bacterial degradation of cyanotoxins microcystins[J]. Toxins, 2016, 8(3): 76. DOI: 10.3390/toxins8030076
[52] Wei J, Huang F Y, Feng H, et al. Characterization and mechanism of linearized-microcystinase involved in bacterial degradation of microcystins[J]. Frontiers in Microbiology, 2021, 12: 646084. DOI: 10.3389/fmicb.2021.646084
[53] 祝晓芸. Rhizobium sp. TH菌降解微囊藻毒素的机理及mlrA基因的进化来源[D]. 武汉:武汉理工大学,2016. [54] Singh D P, Prabha R, Keshri V, et al. Structure prediction and binding site analysis of hepatotoxic microcystin-LR degrading MlrC-like protein from Burkholderia sp. using computational approaches[J]. American Journal of Bioinformatics, 2016, 5(1): 1 − 9.
[55] Wang R P, Li J M, Li J. Functional and structural analyses for MlrC enzyme of Novosphingobium sp. THN1 in microcystin-biodegradation: involving optimized heterologous expression, bioinformatics and site-directed mutagenesis[J]. Chemosphere, 2020, 255: 126906. DOI: 10.1016/j.chemosphere.2020.126906
[56] Okano K, Shimizu K, Maseda H, et al. Whole-genome sequence of the microcystin-degrading bacterium Sphingopyxis sp. strain C-1[J]. Genome Announcements, 2015, 3(4): e00838 − 15.
[57] Jin H Y, Nishizawa T, Guo Y, et al. Complete genome sequence of a microcystin-degrading bacterium, Sphingosinicella microcystinivorans strain B-9[J]. Microbiology Resource Announcements, 2018, 7(9): e00898 − 18.
[58] Jin H Y, Hiraoka Y, Okuma Y, et al. Microbial degradation of amino acid-containing compounds using the microcystin-degrading bacterial strain B-9[J]. Marine Drugs, 2018, 16(2): 50. DOI: 10.3390/md16020050
[59] Yan H, Wang J F, Chen J, et al. Characterization of the first step involved in enzymatic pathway for microcystin-RR biodegraded by Sphingopyxis sp. USTB-05[J]. Chemosphere, 2012, 87(1): 12 − 18. DOI: 10.1016/j.chemosphere.2011.11.030
[60] Saito T, Okano K, Park H D, et al. Detection and sequencing of the microcystin LR-degrading gene, mlrA, from new bacteria isolated from Japanese lakes[J]. FEMS Microbiology Letters, 2003, 229(2): 271 − 276. DOI: 10.1016/S0378-1097(03)00847-4
[61] Krishnan A, Zhang Y Q, Mou X Z. Isolation and characterization of microcystin-degrading bacteria from Lake Erie[J]. Bulletin of Environmental Contamination and Toxicology, 2018, 101(5): 617 − 623. DOI: 10.1007/s00128-018-2468-4
[62] Huang F Y, Feng H, Li X Y, et al. Anaerobic degradation of microcystin-LR by an indigenous bacterial Enterobacter sp. YF3[J]. Journal of Toxicology and Environmental Health, Part A, 2019, 82(21): 1120 − 1128. DOI: 10.1080/15287394.2019.1699345
[63] Qin L, Zhang X X, Chen X G, et al. Isolation of a novel microcystin-degrading bacterium and the evolutionary origin of mlr gene cluster[J]. Toxins, 2019, 11(5): 269. DOI: 10.3390/toxins11050269
[64] Wang R P, Li J M, Jiang Y G, et al. Heterologous expression of mlrA gene originated from Novosphingobium sp. THN1 to degrade microcystin-RR and identify the first step involved in degradation pathway[J]. Chemosphere, 2017, 184: 159 − 167. DOI: 10.1016/j.chemosphere.2017.05.086
[65] Bourne D G, Jones G J, Blakeley R L, et al. Enzymatic pathway for the bacterial degradation of the cyanobacterial cyclic peptide toxin microcystin LR[J]. Applied and Environmental Microbiology, 1996, 62(11): 4086 − 4094. DOI: 10.1128/aem.62.11.4086-4094.1996
[66] Zhang J, Lu Q Q, Ding Q, et al. A novel and native microcystin-degrading bacterium of Sphingopyxis sp. isolated from Lake Taihu[J]. International Journal of Environmental Research and Public Health, 2017, 14(10): 1187. DOI: 10.3390/ijerph14101187
[67] Ding Q, Liu K Y, Xu K, et al. Further understanding of degradation pathways of microcystin-LR by an indigenous Sphingopyxis sp. in environmentally relevant pollution concentrations[J]. Toxins, 2018, 10(12): 536. DOI: 10.3390/toxins10120536
[68] Wang H S, Yan H, Ma S, et al. Characterization of the second and third steps in the enzymatic pathway for microcystin-RR biodegradation by Sphingopyxis sp. USTB-05[J]. Annals of Microbiology, 2015, 65(1): 495 − 502. DOI: 10.1007/s13213-014-0885-0
[69] Xu H M, Wang H S, Xu Q Q, et al. Pathway for biodegrading microcystin-YR by Sphingopyxis sp. USTB-05[J]. PLoS One, 2015, 10(4): e0124425. DOI: 10.1371/journal.pone.0124425
[70] Ding Q, Liu K Y, Song Z Q, et al. Effects of microcystin-LR on metabolic functions and structure succession of sediment bacterial community under anaerobic conditions[J]. Toxins, 2020, 12(3): 183. DOI: 10.3390/toxins12030183
[71] Bao Z Y, Wu Y. Biodegradation of microcystin-LR by an amino acid-degrading anaerobic bacterium[J]. Desalination and Water Treatment, 2016, 57(2): 870 − 880. DOI: 10.1080/19443994.2014.969316
[72] 欧阳潦. CJ5菌降解MCLR的酶学途径研究[D]. 武汉:武汉理工大学,2014. [73] Ding Q, Song X L, Yuan M X, et al. Multiple pathways for the anaerobic biodegradation of microcystin-LR in the enriched microbial communities from Lake Taihu[J]. Environmental Pollution, 2022, 297: 118787. DOI: 10.1016/j.envpol.2022.118787
[74] Zeng Y H, Cai Z H, Zhu J M, et al. Two hierarchical LuxR-LuxI type quorum sensing systems in Novosphingobium activate microcystin degradation through transcriptional regulation of the mlr pathway[J]. Water Research, 2020, 183: 116092. DOI: 10.1016/j.watres.2020.116092
[75] Shen R Y, Chen Z H, Dong X N, et al. Biodegradation kinetics of microcystins-LR crude extract by Lysinibacillus boronitolerans strain CQ5[J]. Annals of Microbiology, 2019, 69(12): 1259 − 1266. DOI: 10.1007/s13213-019-01510-6
[76] Yang F, Zhou Y L, Yin L H, et al. Microcystin-degrading activity of an indigenous bacterial strain Stenotrophomonas acidaminiphila MC-LTH2 isolated from Lake Taihu[J]. PLoS One, 2014, 9(1): e86216. DOI: 10.1371/journal.pone.0086216
[77] Yang F, Huang F Y, Feng H, et al. A complete route for biodegradation of potentially carcinogenic cyanotoxin microcystin-LR in a novel indigenous bacterium[J]. Water Research, 2020, 174: 115638. DOI: 10.1016/j.watres.2020.115638
[78] Zeng Y H, Cheng K K, Cai Z H, et al. Transcriptome analysis expands the potential roles of quorum sensing in biodegradation and physiological responses to microcystin[J]. Science of the Total Environment, 2021, 771: 145437. DOI: 10.1016/j.scitotenv.2021.145437
[79] Idroos F S, De Silva B, Manage P M. Biodegradation of microcystin analogues by Stenotrophomonas maltophilia isolated from Beira Lake Sri Lanka[J]. Journal of the National Science Foundation of Sri Lanka, 2017, 45(2): 91 − 99. DOI: 10.4038/jnsfsr.v45i2.8175
[80] Yang F, Guo J, Huang F Y, et al. Removal of microcystin-LR by a novel native effective bacterial community designated as YFMCD4 isolated from Lake Taihu[J]. Toxins, 2018, 10(9): 363. DOI: 10.3390/toxins10090363
[81] Kang Y H, Park C S, Han M S. Pseudomonas aeruginosa UCBPP-PA14 a useful bacterium capable of lysing Microcystis aeruginosa cells and degrading microcystins[J]. Journal of Applied Phycology, 2012, 24(6): 1517 − 1525. DOI: 10.1007/s10811-012-9812-6
[82] Li H, Ai H N, Kang L, et al. Simultaneous Microcystis algicidal and microcystin degrading capability by a single Acinetobacter bacterial strain[J]. Environmental Science & Technology, 2016, 50(21): 11903 − 11911.
[83] Yu Y, Zeng Y D, Li J, et al. An algicidal Streptomyces amritsarensis strain against Microcystis aeruginosa strongly inhibits microcystin synthesis simultaneou sly[J]. Science of the Total Environment, 2019, 650(Pt 1): 34 − 43.
[84] Pal M, Purohit H J, Qureshi A. Genomic insight for algicidal activity in Rhizobium strain AQ_MP[J]. Archives of Microbiology, 2021, 203(8): 5193 − 5203. DOI: 10.1007/s00203-021-02496-z
[85] Boonbangkeng D, Thiemsorn W, Ruangrit K, et al. Promoting the simultaneous removal of Microcystis bloom and microcystin-RR by Bacillus sp. AK3 immobilized on floating porous glass pellets[J]. Journal of Applied Phycology, 2022, 34(3): 1513 − 1525. DOI: 10.1007/s10811-022-02701-6
[86] Ren G F, He X H, Wu P, et al. Biodegradation of microcystin-RR and nutrient pollutants using Sphingopyxis sp. YF1 immobilized activated carbon fibers-sodium alginate[J]. Environmental Science and Pollution Research, 2020, 27(10): 10811 − 10821. DOI: 10.1007/s11356-020-07640-8
[87] Mawang C I, Azman A S, Fuad A S M, et al. Actinobacteria: an eco-friendly and promising technology for the bioaugmentation of contaminants[J]. Biotechnology Reports, 2021, 32: e00679. DOI: 10.1016/j.btre.2021.e00679
[88] 万翔. 复合降解菌强化人工湿地处理微囊藻毒素LR机制研究[D]. 广州:暨南大学,2017. [89] Wang R, Tai Y P, Wan X, et al. Enhanced removal of Microcystis bloom and microcystin-LR using microcosm constructed wetlands with bioaugmentation of degrading bacteria[J]. Chemosphere, 2018, 210: 29 − 37. DOI: 10.1016/j.chemosphere.2018.06.140
[90] Kumar P, Hegde K, Brar S K, et al. Co-culturing of native bacteria from drinking water treatment plant with known degraders to accelerate microcystin-LR removal using biofilter[J]. Chemical Engineering Journal, 2020, 383: 123090. DOI: 10.1016/j.cej.2019.123090
[91] 王亚军,司运美,李彦娟. 群体感应在生物强化功能菌定殖及降解能力增强中的作用研究进展[J]. 应用生态学报,2022,33(10):2871 − 2880. -
期刊类型引用(1)
1. 林明远. 紫外和过氧化氢去除水中微囊藻毒素和叶绿素的试验研究. 农村科学实验. 2024(14): 160-162 .  百度学术
百度学术
其他类型引用(0)



 下载:
下载: